ДНК и гены
ДНК ПРОКАРИОТ И ЭУКАРИОТ
 |
|
Справа крупнейшая спираль ДНК человека, выстроенная из людей на пляже в Варне (Болгария), вошедшая в книгу рекордов Гиннесса 23 апреля 2016 года
Дезоксирибонуклеиновая кислота. Общие сведения
 Содержание страницы:
Содержание страницы:
- Дезоксирибонуклеиновая кислота
- Строение нуклеиновых кислот
- Репликация
- Строение РНК
- Транскрипция
- Трансляция
- Генетический код
- Геном: гены и хромосомы
- Прокариоты
- Эукариоты
- Строение генов
- Строение генов прокариот
- Строение генов эукариот
- Сравнение строения генов
- Мутации и мутагенез
- Генные мутации
- Хромосомные мутации
- Геномные мутации
- Видео по теме ДНК
- Дополнительный материал
ДНК (дезоксирибонуклеиновая кислота) – своеобразный чертеж жизни, сложный код, в котором заключены данные о наследственной информации. Эта сложная макромолекула способна хранить и передавать наследственную генетическую информацию из поколения в поколение. ДНК определяет такие свойства любого живого организма как наследственность и изменчивость. Закодированная в ней информация задает всю программу развития любого живого организма. Генетически заложенные факторы предопределяют весь ход жизни как человека, так и любого др. организхма. Искусственное или естественное воздействие внешней среды способны лишь в незначительной степени повлиять на общую выраженность отдельных генетических признаков или сказаться на развитии запрограммированных процессов.
Дезоксирибонуклеи́новая кислота (ДНК) — макромолекула (одна из трёх основных, две другие — РНК и белки), обеспечивающая хранение, передачу из поколения в поколение и реализацию генетической программы развития и функционирования живых организмов. ДНК содержит информацию о структуре различных видов РНК и белков.
В клетках эукариот (животных, растений и грибов) ДНК находится в ядре клетки в составе хромосом, а также в некоторых клеточных органоидах (митохондриях и пластидах). В клетках прокариотических организмов (бактерий и архей) кольцевая или линейная молекула ДНК, так называемый нуклеоид, прикреплена изнутри к клеточной мембране. У них и у низших эукариот (например, дрожжей) встречаются также небольшие автономные, преимущественно кольцевые молекулы ДНК, называемые плазмидами.
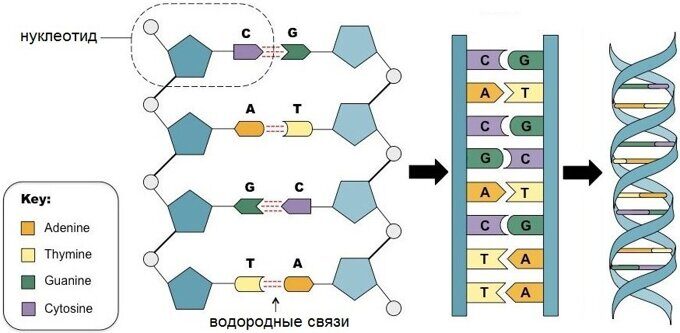
С химической точки зрения ДНК — это длинная полимерная молекула, состоящая из повторяющихся блоков — нуклеотидов. Каждый нуклеотид состоит из азотистого основания, сахара (дезоксирибозы) и фосфатной группы. Связи между нуклеотидами в цепи образуются за счёт дезоксирибозы (С) и фосфатной (Ф) группы (фосфодиэфирные связи).

Рис. 2. Нуклертид состоит из азотистого основания, сахара (дезоксирибозы) и фосфатной группы
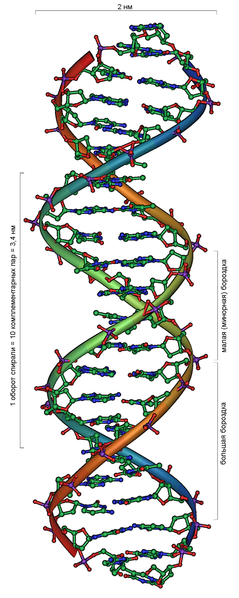
В подавляющем большинстве случаев (кроме некоторых вирусов, содержащих одноцепочечную ДНК) макромолекула ДНК состоит из двух цепей, ориентированных азотистыми основаниями друг к другу. Эта двухцепочечная молекула закручена по винтовой линии.
В ДНК встречается четыре вида азотистых оснований (аденин, гуанин, тимин и цитозин). Азотистые основания одной из цепей соединены с азотистыми основаниями другой цепи водородными связями согласно принципу комплементарности: аденин соединяется только с тимином (А-Т), гуанин — только с цитозином (Г-Ц). Именно эти пары и составляют «перекладины» винтовой «лестницы» ДНК (см.: рис. 2, 3 и 4).

Рис. 2. Азотистые основания
Последовательность нуклеотидов позволяет «кодировать» информацию о различных типах РНК, наиболее важными из которых являются информационные, или матричные (мРНК), рибосомальные (рРНК) и транспортные (тРНК). Все эти типы РНК синтезируются на матрице ДНК за счёт копирования последовательности ДНК в последовательность РНК, синтезируемой в процессе транскрипции, и принимают участие в биосинтезе белков (процессе трансляции). Помимо кодирующих последовательностей, ДНК клеток содержит последовательности, выполняющие регуляторные и структурные функции.

Рис. 3. Репликация ДНК
Расположение базовых комбинаций химических соединений ДНК и количественные соотношения между этими комбинациями обеспечивают кодирование наследственной информации.
Образование новой ДНК (репликация)
- Процесс репликации: раскручивание двойной спирали ДНК — синтез комплементарных цепей ДНК-полимеразой — образование двух молекул ДНК из одной.
- Двойная спираль «расстегивается» на две ветви, когда ферменты разрушают связь между базовыми парами химических соединений.
- Каждая ветвь является элементом новой ДНК. Новые базовые пары соединяются в той же последовательности, что и в родительской ветви.
По завершении дупликации образуются две самостоятельные спирали, созданные из химических соединений родительской ДНК и имеющие с ней одинаковый генетический код. Таким путем ДНК способна перерывать информацию от клетки к клетке.
Более подробная информация:
СТРОЕНИЕ НУКЛЕИНОВЫХ КИСЛОТ

Рис. 4 . Азотистые основания: аденин, гуанин, цитозин, тимин
Дезоксирибонуклеиновая кислота (ДНК) относится к нуклеиновым кислотам. Нуклеиновые кислоты – это класс нерегулярных биополимеров, мономерами которых являются нуклеотиды.
НУКЛЕОТИДЫ состоят из азотистого основания, соединенного с пятиуглеродным углеводом (пентозой) – дезоксирибозой (в случае ДНК) или рибозой (в случае РНК), который соединяется с остатком фосфорной кислоты (H2PO3–).
Азотистые основания бывают двух типов: пиримидиновые основания – урацил (только в РНК), цитозин и тимин, пуриновые основания – аденин и гуанин.

Рис. 5. Структура нуклеотидов (слева), расположение нуклеотида в ДНК (снизу) и типы азотистых оснований (справа): пиримидиновые и пуриновые
Атомы углерода в молекуле пентозы нумеруются числами от 1 до 5. Фосфат соединяется с третьим и пятым атомами углерода. Так нуклеинотиды соединяются в цепь нуклеиновой кислоты. Таким образом, мы можем выделить 3’ и 5’-концы цепи ДНК:
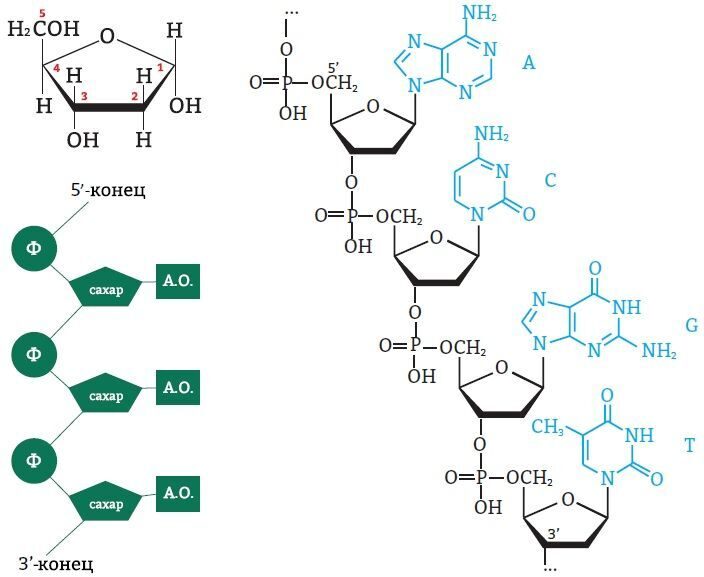
Рис. 6. Выделение 3’ и 5’-концов цепи ДНК
Две цепи ДНК образуют двойную спираль. Эти цепи в спирали сориентированы в противоположных направлениях. В разных цепях ДНК азотистые основания соединены между собой с помощью водородных связей. Аденин всегда соединяется с тимином, а цитозин – с гуанином. Это называется правилом комплементарности (см. принцип комплементарности).
Правило комплементарности:
Например, если нам дана цепь ДНК, имеющая последовательность
3’– ATGTCCTAGCTGCTCG – 5’,
то вторая ей цепь будет комплементарна и направлена в противоположном направлении – от 5’-конца к 3’-концу:
5’– TACAGGATCGACGAGC– 3’.

Рис. 7. Направленность цепей молекулы ДНК и соединение азотистых оснований с помощью водородных связей
РЕПЛИКАЦИЯ ДНК
Репликация ДНК – это процесс удвоения молекулы ДНК путем матричного синтеза. В большинстве случаев естественной репликации ДНК праймером для синтеза ДНК является короткий фрагмент РНК (создаваемый заново). Такой рибонуклеотидный праймер создается ферментом праймазой (ДНК-праймаза у прокариот, ДНК-полимераза у эукариот), и впоследствии заменяется дезоксирибонуклеотидами полимеразой, выполняющей в норме функции репарации (исправления химических повреждений и разрывов в молекле ДНК).
Репликация происходит по полуконсервативному механизму. Это значит, что двойная спираль ДНК расплетается и на каждой из ее цепей по принципу комплементарности достраивается новая цепь. Дочерняя молекула ДНК, таким образом, содержит в себе одну цепь от материнской молекулы и одну вновь синтезированную. Репликация происходит в направлении от 3’ к 5’ концу материнской цепи.

Рис. 8. Репликация (удвоение) молекулы ДНК
ДНК-синтез – это не такой сложный процесс, как может показаться на первый взгляд. Если подумать, то для начала нужно разобраться, что же такое синтез. Это процесс объединения чего-либо в одно целое. Образование новой молекулы ДНК проходит в несколько этапов:
1) ДНК-топоизомераза, располагаясь перед вилкой репликации, разрезает ДНК для того, чтобы облегчить ее расплетание и раскручивание.
2) ДНК-хеликаза вслед за топоизомеразой влияет на процесс «расплетения» спирали ДНК.
3) ДНК-связывающие белки осуществляют связывание нитей ДНК, а также проводят их стабилизацию, не допуская их прилипания друг к другу.
4) ДНК-полимераза δ (дельта), согласовано со скоростью движения репликативной вилки, осуществляет синтез ведущей цепи дочерней ДНК в направлении 5’→3′ на матрице материнской нити ДНК по направлению от ее 3′-конца к 5′-концу (скорость до 100 пар нуклеотидов в секунду). Этим события на данной материнской нити ДНК ограничиваются.

Рис. 9. Схематическое изображение процесса репликации ДНК: (1) Отстающая цепь (запаздывающая нить), (2) Ведущая цепь (лидирующая нить), (3) ДНК-полимераза α (Polα), (4) ДНК-лигаза, (5) РНК-праймер, (6) Праймаза, (7) Фрагмент Оказаки, (8) ДНК-полимераза δ (Polδ), (9) Хеликаза, (10) Однонитевые ДНК-связывающие белки, (11) Топоизомераза.
Далее описан синтез отстающей цепи дочерней ДНК (см. Схему репликативной вилки и функции ферментов репликации)
Нагляднее о репликации ДНК см. видео →
5) Непосредственно сразу после расплетания и стабилизации другой нити материнской молекулы к ней присоединяется ДНК-полимераза α (альфа) и в направлении 5’→3′ синтезирует праймер (РНК-затравку) – последовательность РНК на матрице ДНК длиной от 10 до 200 нуклеотидов. После этого фермент удаляется с нити ДНК.
Вместо ДНК-полимеразы α к 3′-концу праймера присоединяется ДНК-полимераза ε.
6) ДНК-полимераза ε (эпсилон) как бы продолжает удлинять праймер, но в качестве субстрата встраивает дезоксирибонуклеотиды (в количестве 150-200 нуклеотидов). В результате образуется цельная нить из двух частей – РНК (т.е. праймер) и ДНК. ДНК-полимераза ε работает до тех пор, пока не встретит праймер предыдущего фрагмента Оказаки (синтезированный чуть ранее). После этого данный фермент удаляется с цепи.
7) ДНК-полимераза β (бета) встает вместо ДНК-полимеразы ε, движется в том же направлении (5’→3′) и удаляет рибонуклеотиды праймера, одновременно встраивая дезоксирибонуклеотиды на их место. Фермент работает до полного удаления праймера, т.е. пока на его пути не встанет дезоксирибонуклеотид (еще более ранее синтезированный ДНК-полимеразой ε). Связать результат свой работы и впереди стоящую ДНК фермент не в состоянии, поэтому он сходит с цепи.
В результате на матрице материнской нити «лежит» фрагмент дочерней ДНК. Он называется фрагмент Оказаки.
ДНК-лигаза производит сшивку двух соседних фрагментов Оказаки, т.е. 5′-конца отрезка, синтезированного ДНК-полимеразой ε, и 3′-конца цепи, встроенного ДНК-полимеразой β.
СТРОЕНИЕ РНК
Рибонуклеиновая кислота (РНК) — одна из трёх основных макромолекул (две другие — ДНК и белки), которые содержатся в клетках всех живых организмов.
Так же, как ДНК, РНК состоит из длинной цепи, в которой каждое звено называется нуклеотидом. Каждый нуклеотид состоит из азотистого основания, сахара рибозы и фосфатной группы. Однако в отличие от ДНК, РНК обычно имеет не две цепи, а одну. Пентоза в РНК представлена рибозой, а не дезоксирибозой (у рибозы присутствует дополнительная гидроксильная группа на втором атоме углевода). Наконец, ДНК отличается от РНК по составу азотистых оснований: вместо тимина (Т) в РНК представлен урацил (U), который также комплементарен аденину.
Последовательность нуклеотидов позволяет РНК кодировать генетическую информацию. Все клеточные организмы используют РНК (мРНК) для программирования синтеза белков.
Клеточные РНК образуются в ходе процесса, называемого транскрипцией, то есть синтеза РНК на матрице ДНК, осуществляемого специальными ферментами — РНК-полимеразами.
Затем матричные РНК (мРНК) принимают участие в процессе, называемом трансляцией, т.е. синтеза белка на матрице мРНК при участии рибосом. Другие РНК после транскрипции подвергаются химическим модификациям, и после образования вторичной и третичной структур выполняют функции, зависящие от типа РНК.

Рис. 10. Отличие ДНК от РНК по азотистому основанию: вместо тимина (Т) в РНК представлен урацил (U), который также комплементарен аденину.
ТРАНСКРИПЦИЯ
Транскрипция – это процесс синтеза РНК на матрице ДНК. ДНК раскручивается на одном из участков. На одной из цепей содержится информация, которую необходимо скопировать на молекулу РНК – эта цепь называется кодирующей. Вторая цепь ДНК, комплементарная кодирующей, называется матричной. В процессе транскрипции на матричной цепи в направлении 3’ – 5’ (по цепи ДНК) синтезируется комплементарная ей цепь РНК. Таким образом, создается РНК-копия кодирующей цепи.

Рис. 11. Схематическое изображение транскрипции
Например, если нам дана последовательность кодирующей цепи
3’– ATGTCCTAGCTGCTCG – 5’,
то, по правилу комплементарности, матричная цепь будет нести последовательность
5’– TACAGGATCGACGAGC– 3’,
а синтезируемая с нее РНК – последовательность
3’– AUGUCCUAGCUGCUCG – 5’.
ТРАНСЛЯЦИЯ
Рассмотрим механизм синтеза белка на матрице РНК, а также генетический код и его свойства. Также для наглядности по ниже приведенной ссылке рекомендуем посмотреть небольшое видео о процессах транскрипции и трансляции, происходящих в живой клетке:
|
|
В представленном видоролике (кнопка-ссылка слева) показан процесс образования белка из аминокислот. Наглядно (в анимированном варианте) продемонстрированы процессы транскрипции и трансляции. Биосинтез белка на рибосоме также кратко описан в разделе Аминокислоты белков. Более подробное видео о геноме, ДНК и ее структуре, а также процессах кодировки представленно ниже на данной странице: Видео по теме ДНК |

Рис. 12. Процесс синтеза белка: ДНК кодирует РНК, РНК кодирует белок
Трансляция — это процесс, посредством которого генетическая информация преобразуется в белки, рабочие лошадки клетки. Небольшие молекулы, называемые переносными РНК («тРНК»), играют решающую роль в трансляции; они являются молекулами-адаптерами, которые соответствуют кодонам (строительным блокам генетической информации) с аминокислотами (строительными блоками белков). Организмы несут множество типов тРНК, каждая из которых кодируется одним или несколькими генами («набор генов тРНК»).
Вообще говоря, функция набора генов тРНК — переводить 61 тип кодонов в 20 различных типов аминокислот — сохраняется в разных организмах. Тем не менее, состав набора генов тРНК может значительно варьировать между организмами.
ГЕНЕТИЧЕСКИЙ КОД
Генетический код — способ кодирования аминокислотной последовательности белков с помощью последовательности нуклеотидов. Каждая аминокислота кодируется последовательностью из трех нуклеотидов — кодоном или триплетом.
Генетический код, общий для большинства про- и эукариот. В таблице приведены все 64 кодона и указаны соответствующие аминокислоты. Порядок оснований — от 5′ к 3′ концу мРНК.
Таблица 1. Стандартный генетический код
|
1-е ние |
2-е основание |
3-е ние |
|||||||
|
U |
C |
A |
G |
||||||
|
U |
UUU |
Фенилаланин (Phe/F) |
UCU |
Серин (Ser/S) |
UAU |
Тирозин (Tyr/Y) |
UGU |
Цистеин (Cys/C) |
U |
|
UUC |
UCC |
UAC |
UGC |
C |
|||||
|
UUA |
Лейцин (Leu/L) |
UCA |
UAA |
Стоп-кодон** |
UGA |
Стоп-кодон** |
A |
||
|
UUG |
UCG |
UAG |
Стоп-кодон** |
UGG |
Триптофан (Trp/W) |
G |
|||
|
C |
CUU |
CCU |
Пролин (Pro/P) |
CAU |
Гистидин (His/H) |
CGU |
Аргинин (Arg/R) |
U |
|
|
CUC |
CCC |
CAC |
CGC |
C |
|||||
|
CUA |
CCA |
CAA |
Глутамин (Gln/Q) |
CGA |
A |
||||
|
CUG |
CCG |
CAG |
CGG |
G |
|||||
|
A |
AUU |
Изолейцин (Ile/I) |
ACU |
Треонин (Thr/T) |
AAU |
Аспарагин (Asn/N) |
AGU |
Серин (Ser/S) |
U |
|
AUC |
ACC |
AAC |
AGC |
C |
|||||
|
AUA |
ACA |
AAA |
Лизин (Lys/K) |
AGA |
Аргинин (Arg/R) |
A |
|||
|
AUG |
Метионин* (Met/M) |
ACG |
AAG |
AGG |
G |
||||
|
G |
GUU |
Валин (Val/V) |
GCU |
Аланин (Ala/A) |
GAU |
Аспарагиновая кислота (Asp/D) |
GGU |
Глицин (Gly/G) |
U |
|
GUC |
GCC |
GAC |
GGC |
C |
|||||
|
GUA |
GCA |
GAA |
Глутаминовая кислота (Glu/E) |
GGA |
A |
||||
|
GUG |
GCG |
GAG |
GGG |
G |
Среди триплетов есть 4 специальных последовательности, выполняющих функции «знаков препинания»:
- *Триплет AUG, также кодирующий метионин, называется старт-кодоном. С этого кодона начинается синтез молекулы белка. Таким образом, во время синтеза белка, первой аминокислотой в последовательности всегда будет метионин.
- **Триплеты UAA, UAG и UGA называются стоп-кодонами и не кодируют ни одной аминокислоты. На этих последовательностях синтез белка прекращается.
Свойства генетического кода
1. Триплетность. Каждая аминокислота кодируется последовательностью из трех нуклеотидов – триплетом или кодоном.

2. Непрерывность. Между триплетами нет никаких дополнительных нуклеотидов, информация считывается непрерывно.

3. Неперекрываемость. Один нуклеотид не может входить одновременно в два триплета.

4. Однозначность. Один кодон может кодировать только одну аминокислоту.

5. Вырожденность. Одна аминокислота может кодироваться несколькими разными кодонами.

6. Универсальность. Генетический код одинаков для всех живых организмов.
Пример. Нам дана последовательность кодирующей цепи:
3’– CCGATTGCACGTCGATCGTATA– 5’.
Матричная цепь будет иметь последовательность:
5’– GGCTAACGTGCAGCTAGCATAT– 3’.
Теперь «синтезируем» с этой цепи информационную РНК:
3’– CCGAUUGCACGUCGAUCGUAUA– 5’.
Синтез белка идет в направлении 5’ → 3’, следовательно, нам нужно перевернуть последовательность, чтобы «прочитать» генетический код:
5’– AUAUGCUAGCUGCACGUUAGCC– 3’.
Теперь найдем старт-кодон AUG:
5’– AUAUGCUAGCUGCACGUUAGCC– 3’.
Разделим последовательность на триплеты:
![]()
Найдем стоп-кодон и согласно таблице генетического кода запишем последовательность аминокислот:

Центральная догма молекулярной биологии звучит следующим образом: информация с ДНК передается на РНК (транскрипция), с РНК – на белок (трансляция). ДНК также может удваиваться путем репликации, и также возможен процесс обратной транскрипции, когда по матрице РНК синтезируется ДНК, но такой процесс в основном характерен для вирусов.

Рис. 13. Центральная догма молекулярной биологии
ГЕНОМ: ГЕНЫ и ХРОМОСОМЫ
(общие понятия)
Геном — совокупность всех генов организма; его полный хромосомный набор.
Термин «геном» был предложен Г. Винклером в 1920 г. для описания совокупности генов, заключенных в гаплоидном наборе хромосом организмов одного биологического вида. Первоначальный смысл этого термина указывал на то, что понятие генома в отличие от генотипа является генетической характеристикой вида в целом, а не отдельной особи. С развитием молекулярной генетики значение данного термина изменилось. Известно, что ДНК, которая является носителем генетической информации у большинства организмов и, следовательно, составляет основу генома, включает в себя не только гены в современном смысле этого слова. Большая часть ДНК эукариотических клеток представлена некодирующими («избыточными») последовательностями нуклеотидов, которые не заключают в себе информации о белках и нуклеиновых кислотах. Таким образом, основную часть генома любого организма составляет вся ДНК его гаплоидного набора хромосом.
Гены — это участки молекул ДНК, кодирующие полипептиды и молекулы РНК
За последнее столетие наше представление о генах существенно изменилось. Ранее геном называли участок хромосомы, кодирующий или определяющий один признак или фенотипическое (видимое) свойство, например цвет глаз.
|
|
|
Рис. 14. Соответствие между кодирующими участками ДНК, мРНК и аминокислотной последовательностью полипептидной цепи. |
В 1940 г. Джордж Бидл и Эдвард Тейтем предложили молекулярное определение гена. Ученые обрабатывали споры гриба Neurospora crassa рентгеновским излучением и другими агентами, вызывающими изменения в последовательности ДНК (мутации), и обнаружили мутантные штаммы гриба, утратившие некоторые специфические ферменты, что в некоторых случаях приводило к нарушению целого метаболического пути. Бидл и Тейтем пришли к выводу, что ген — это участок генетического материала, который определяет или кодирует один фермент. Так появилась гипотеза «один ген — один фермент». Позднее эта концепция была расширена до определения «один ген — один полипептид», поскольку многие гены кодируют белки, не являющиеся ферментами, а полипептид может оказаться субъединицей сложного белкового комплекса.
На рис. 14 показана схема того, как триплеты нуклеотидов в ДНК определяют полипептид — аминокислотную последовательность белка при посредничестве мРНК. Одна из цепей ДНК играет роль матрицы для синтеза мРНК, нуклеотидные триплеты (кодоны) которой комплементарны триплетам ДНК. У некоторых бактерий и многих эукариот кодирующие последовательности прерываются некодирующими участками(так называемыми интронами).
Современное биохимическое определение гена еще более конкретно. Генами называются все участки ДНК, кодирующие первичную последовательность конечных продуктов, к которым относятся полипептиды или РНК, обладающие структурной или каталитической функцией.
Наряду с генами ДНК содержит и другие последовательности, выполняющие исключительно регуляторную функцию. Регуляторные последовательности могут обозначать начало или конец генов, влиять на транскрипцию или указывать место инициации репликации или рекомбинации. Некоторые гены могут экспрессироваться разными путями, при этом один и тот же участок ДНК служит матрицей для образования разных продуктов.
Мы можем приблизительно рассчитать минимальный размер гена, кодирующего средний белок. Каждая аминокислота в полипептидной цепи кодируется последовательностью из трех нуклеотидов; последовательности этих триплетов (кодонов) соответствуют цепочке аминокислот в полипептиде, который кодируется данным геном. Полипептидная цепь из 350 аминокислотных остатков (цепь средней длины) соответствует последовательности из 1050 п.н. (пар нуклеотидов). Однако многие гены эукариот и некоторые гены прокариот прерываются сегментами ДНК, не несущими информации о белке, и поэтому оказываются значительно длиннее, чем показывает простой расчет.
Сколько генов в одной хромосоме?
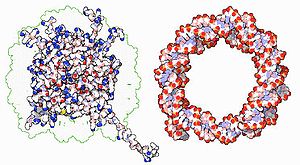
 Рис. 15. Вид хромосом в прокаритической (слева) и эукариотической клеках. Гистоны (Histones) — обширный класс ядерных белков, выполняющих две основные функции: они участвуют в упаковке нитей ДНК в ядре и в эпигенетической регуляции таких ядерных процессов, как транскрипция, репликация и репарация.
Рис. 15. Вид хромосом в прокаритической (слева) и эукариотической клеках. Гистоны (Histones) — обширный класс ядерных белков, выполняющих две основные функции: они участвуют в упаковке нитей ДНК в ядре и в эпигенетической регуляции таких ядерных процессов, как транскрипция, репликация и репарация.
ДНК прокариот устроена более просто: их клетки не имеют ядра, поэтому ДНК находится непосредственно в цитоплазме в форме нуклеоида.
 Как известно, бактериальные клетки имеют хромосому в виде нити ДНК, уложенной в компактную структуру – нуклеоид. Хромосома прокариота Escherichia coli, чей геном полностью расшифрован, представляет собой кольцевую молекулу ДНК (на самом деле, это не правильный круг, а скорее петля без начала и конца), состоящую из 4 639 675 п.н. В этой последовательности содержится примерно 4300 генов белков и еще 157 генов стабильных молекул РНК. В геноме человека примерно 3,1 млрд пар нуклеотидов, соответствующих почти 29 000 генам, расположенным на 24 разных хромосомах.
Как известно, бактериальные клетки имеют хромосому в виде нити ДНК, уложенной в компактную структуру – нуклеоид. Хромосома прокариота Escherichia coli, чей геном полностью расшифрован, представляет собой кольцевую молекулу ДНК (на самом деле, это не правильный круг, а скорее петля без начала и конца), состоящую из 4 639 675 п.н. В этой последовательности содержится примерно 4300 генов белков и еще 157 генов стабильных молекул РНК. В геноме человека примерно 3,1 млрд пар нуклеотидов, соответствующих почти 29 000 генам, расположенным на 24 разных хромосомах.
Прокариоты (Бактерии).
 Бактерия E. coli имеет одну двухцепочечную кольцевую молекулу ДНК. Она состоит из 4 639 675 п.н. и достигает в длину примерно 1,7 мм, что превышает длину самой клетки E. coli приблизительно в 850 раз. Помимо крупной кольцевой хромосомы в составе нуклеоида многие бактерии содержат одну или несколько маленьких кольцевых молекул ДНК, свободно располагающихся в цитозоле. Такие внехромосомные элементы называют плазмидами (рис. 16).
Бактерия E. coli имеет одну двухцепочечную кольцевую молекулу ДНК. Она состоит из 4 639 675 п.н. и достигает в длину примерно 1,7 мм, что превышает длину самой клетки E. coli приблизительно в 850 раз. Помимо крупной кольцевой хромосомы в составе нуклеоида многие бактерии содержат одну или несколько маленьких кольцевых молекул ДНК, свободно располагающихся в цитозоле. Такие внехромосомные элементы называют плазмидами (рис. 16).
Большинство плазмид состоит всего из нескольких тысяч пар нуклеотидов, некоторые содержат более 10000 п. н. Они несут генетическую информацию и реплицируются с образованием дочерних плазмид, которые попадают в дочерние клетки в процессе деления родительской клетки. Плазмиды обнаружены не только в бактериях, но также в дрожжах и других грибах. Во многих случаях плазмиды не дают никаких преимуществ клеткам-хозяевам, и их единственная задача — независимое воспроизведение. Однако некоторые плазмиды несут полезные для хозяина гены. Например, содержащиеся в плазмидах гены могут придавать клеткам бактерий устойчивость к антибактериальным агентам. Плазмиды, несущие ген β-лактамазы, обеспечивают устойчивость к β-лактамным антибиотикам, таким как пенициллин и амоксициллин. Плазмиды могут переходить от клеток, устойчивых к антибиотикам, к другим клеткам того же или другого вида бактерий, в результате чего эти клетки также становятся резистентными. Интенсивное применение антибиотиков является мощным селективным фактором, способствующим распространению плазмид, кодирующих устойчивость к антибиотикам (а также транспозонов, которые кодируют аналогичные гены) среди болезнетворных бактерий, и приводит к появлению бактериальных штаммов с устойчивостью к нескольким антибиотикам. Врачи начинают понимать опасность широкого использования антибиотиков и назначают их только в случае острой необходимости. По аналогичным причинам ограничивается широкое использование антибиотиков для лечения сельскохозяйственных животных.
См. также: Равин Н.В., Шестаков С.В. Геном прокариот // Вавиловский журнал генетики и селекции, 2013. Т. 17. № 4/2. С. 972–984.
Эукариоты.
Таблица 2. ДНК, гены и хромосомы некоторых организмов
|
Общая ДНК, п.н. |
Число хромосом* |
Примерное число генов |
|
|
Escherichia coli (бактерия) |
4 639 675 |
1 |
4 435 |
|
Saccharomyces cerevisiae (дрожжи) |
12 080 000 |
16** |
5 860 |
|
Caenorhabditis elegans (нематода) |
90 269 800 |
12*** |
23 000 |
|
Arabidopsis thaliana (растение) |
119 186 200 |
10 |
33 000 |
|
Drosophila melanogaster (плодовая мушка) |
120 367 260 |
18 |
20 000 |
|
Oryza sativa (рис) |
480 000 000 |
24 |
57 000 |
|
Mus musculus (мышь) |
2 634 266 500 |
40 |
27 000 |
|
Homo sapiens (человек) |
3 070 128 600 |
46 |
29 000 |
Примечание. Информация постоянно обновляется; для получения более свежей информации обратитесь к сайтам, посвященным отдельным геномным проектам
*Для всех эукариот, кроме дрожжей, приводится диплоидный набор хромосом. Диплоидный набор хромосом (от греч. diploos- двойной и eidos- вид) – двойной набор хромосом (2n), каждая из которых имеет себе гомологичную.
**Гаплоидный набор. Дикие штаммы дрожжей обычно имеют восемь (октаплоидный) или больше наборов таких хромосом.
***Для самок с двумя Х хромосомами. У самцов есть Х хромосома, но нет Y, т. е. всего 11 хромосом.
В клетке дрожжей, одних из самых маленьких эукариот, в 2,6 раза больше ДНК, чем в клетке E. coli (табл. 2). Клетки плодовой мушки Drosophila, классического объекта генетических исследований, содержат в 35 раз больше ДНК, а клетки человека — примерно в 700 раз больше ДНК, чем клетки E. coli. Многие растения и амфибии содержат еще больше ДНК. Генетический материал клеток эукариот организован в виде хромосом. Диплоидный набор хромосом (2n) зависит от вида организма (табл. 2).
Например, в соматической клетке человека 46 хромосом (рис. 17). Каждая хромосома эукариотической клетки, как показано на рис. 17, а, содержит одну очень крупную двухспиральную молекулу ДНК. Двадцать четыре хромосомы человека (22 парные хромосомы и две половые хромосомы X и Y) различаются по длине более чем в 25 раз. Каждая хромосома эукариот содержит определенный набор генов.

Рис. 17. Хромосомы эукариот. а — пара связанных и конденсированных сестринских хроматид из хромосомы человека. В такой форме эукариотические хромосомы пребывают после репликации и в метафазе в процессе митоза. б — полный набор хромосом из лейкоцита одного из авторов книги. В каждой нормальной соматической клетке человека содержится 46 хромосом.

Размер и функция ДНК как матрицы для хранения и передачи наследственного материала объясняют наличие особых структурных элементов в организации этой молекулы. У высших организмов ДНК распределена между хромосомами.
Совокупность ДНК (хромосом) организма называется геномом. Хромосомы находятся в клеточном ядре и формируют структуру, называемую хроматином. Хроматин представляет собой комплекс ДНК и основных белков (гистонов) в соотношении 1:1. Длину ДНК обычно измеряют числом пар комплементарных нуклеотидов (п.н.). Например, 3-я хромосома человека представляет собой молекулу ДНК размером 160 млн п.н.. Выделенная линеаризованная ДНК размером 3*106 п.н. имеет длину примерно 1 мм, следовательно, линеаризованная молекула 3-й хромосомы человека была бы 5 мм в длину, а ДНК всех 23 хромосом (~3*109 п.н., MR = 1,8*1012) гаплоидной клетки – яйцеклетки или сперматозоида – в линеаризованном виде составляла бы 1 м. За исключением половых клеток, все клетки организма человека (их около 1013) содержат двойной набор хромосом. При клеточном делении все 46 молекул ДНК реплицируются и снова организуются в 46 хромосом.
Если соединить между собой молекулы ДНК человеческого генома (22 хромосомы и хромосомы X и Y или Х и Х), получится последовательность длиной около одного метра. Прим.: У всех млекопитающих и других организмов с гетерогаметным мужским полом, у самок две X-хромосомы (XX), а у самцов — одна X-хромосома и одна Y-хромосома (XY).
Большинство клеток человека диплоидны, поэтому общая длина ДНК таких клеток около 2м. У взрослого человека примерно 1014 клеток, таким образом, общая длина всех молекул ДНК составляет 2・1011 км. Для сравнения, окружность Земли — 4・104 км, а расстояние от Земли до Солнца — 1,5・108 км. Вот как удивительно компактно упакована ДНК в наших клетках!
В клетках эукариот есть и другие органеллы, содержащие ДНК, — это митохондрии и хлоропласты. Выдвигалось множество гипотез относительно происхождения ДНК митохондрий и хлоропластов. Общепризнанная сегодня точка зрения заключается в том, что они представляют собой рудименты хромосом древних бактерий, которые проникли в цитоплазму хозяйских клеток и стали предшественниками этих органелл. Митохондриальная ДНК кодирует митохондриальные тРНК и рРНК, а также несколько митохондриальных белков. Более 95% митохондриальных белков кодируется ядерной ДНК.
СТРОЕНИЕ ГЕНОВ
Рассмотрим строение гена у прокариот и эукариот, их сходства и различия. Несмотря на то, что ген — это участок ДНК, кодирующий всего один белок или РНК, кроме непосредственно кодирующей части, он также включает в себя регуляторные и иные структурные элементы, имеющие разное строение у прокариот и эукариот.
Кодирующая последовательность – основная структурно-функциональная единица гена, именно в ней находятся триплеты нуклеотидов, кодирующие аминокислотную последовательность. Она начинается со старт-кодона и заканчивается стоп-кодоном.
До и после кодирующей последовательности находятся нетранслируемые 5’- и 3’-последовательности. Они выполняют регуляторные и вспомогательные функции, например, обеспечивают посадку рибосомы на и-РНК.
Нетранслируемые и кодирующая последовательности составлют единицу транскрипции – транскрибируемый участок ДНК, то есть участок ДНК, с которого происходит синтез и-РНК.
Терминатор – нетранскрибируемый участок ДНК в конце гена, на котором останавливается синтез РНК.
В начале гена находится регуляторная область, включающая в себя промотор и оператор.
Промотор – последовательность, с которой связывается полимераза в процессе инициации транскрипции. Оператор – это область, с которой могут связываться специальные белки – репрессоры, которые могут уменьшать активность синтеза РНК с этого гена – иначе говоря, уменьшать его экспрессию.
Строение генов у прокариот
Общий план строения генов у прокариот и эукариот не отличается – и те, и другие содержат регуляторную область с промотором и оператором, единицу транскрипции с кодирующей и нетранслируемыми последовательностями и терминатор. Однако организация генов у прокариот и эукариот отличается.

Рис. 18. Схема строения гена у прокариот (бактерий) — изображение увеличивается
В начале и в конце оперона есть единые регуляторные области для нескольких структурных генов. С транскрибируемого участка оперона считывается одна молекула и-РНК, которая содержит несколько кодирующих последовательностей, в каждой из которых есть свой старт- и стоп-кодон. С каждого из таких участков синтезируется один белок. Таким образом, с одной молекулы и-РНК синтезируется несколько молекул белка.
Для прокариот характерно объединение нескольких генов в единую функциональную единицу – оперон. Работу оперона могут регулировать другие гены, которые могут быть заметно удалены от самого оперона – регуляторы. Белок, транслируемый с этого гена называется репрессор. Он связывается с оператором оперона, регулируя экспрессию сразу всех генов, в нем содержащихся.
Для прокариот также характерно явление сопряжения транскрипции и трансляции.

Рис. 19 Явление сопряжения транскрипции и трансляции у прокариот — изображение увеличивается
Такое сопряжение не встречается у эукариот из-за наличия у них ядерной оболочки, отделяющей цитоплазму, где происходит трансляция, от генетического материала, на котором происходит транскрипция. У прокариот во время синтеза РНК на матрице ДНК с синтезируемой молекулой РНК может сразу связываться рибосома. Таким образом, трансляция начинается еще до завершения транскрипции. Более того, с одной молекулой РНК может одновременно связываться несколько рибосом, синтезируя сразу несколько молекул одного белка.
Строение генов у эукариот
Гены и хромосомы эукариот очень сложно организованы
У бактерий многих видов всего одна хромосома, и почти во всех случаях в каждой хромосоме присутствует по одной копии каждого гена. Лишь немногие гены, например гены рРНК, содержатся в нескольких копиях. Гены и регуляторные последовательности составляют практически весь геном прокариот. Более того, почти каждый ген строго соответствует аминокислотной последовательности (или последовательности РНК), которую он кодирует (рис. 14).
Структурная и функциональная организация генов эукариот гораздо сложнее. Исследование хромосом эукариот, а позднее секвенирование полных последовательностей геномов эукариот принесло много сюрпризов. Многие, если не большинство, генов эукариот обладают интересной особенностью: их нуклеотидные последовательности содержат один или несколько участков ДНК, в которых не кодируется аминокислотная последовательность полипептидного продукта. Такие нетранслируемые вставки нарушают прямое соответствие между нуклеотидной последовательностью гена и аминокислотной последовательностью кодируемого полипептида. Эти нетранслируемые сегменты в составе генов называют интронами, или встроенными последовательностями, а кодирующие сегменты — экзонами. У прокариот лишь немногие гены содержат интроны.
Итак, у эукариот практически не встречается объединение генов в опероны, и кодирующая последовательность гена эукариот чаще всего разделена на транслируемые участки – экзоны, и нетранслируемые участки – интроны.
В большинстве случаев функция интронов не установлена. В целом, лишь около 1,5% ДНК человека являются ≪кодирующими≫, т. е. несут информацию о белках или РНК. Однако с учетом крупных интронов получается, что ДНК человека на 30% состоит из генов. Поскольку гены составляют относительно небольшую долю в геноме человека, значительная часть ДНК остается неучтенной.

Рис. 16. Схема строение гена у эукариот — изображение увеличивается
С каждого гена сначала синтезируется незрелая, или пре-РНК, которая содержит в себе как интроны, так и экзоны.
После этого проходит процесс сплайсинга, в результате которого интронные участки вырезаются, и образуется зрелая иРНК, с которой может быть синтезирован белок.

Рис. 20. Процесс альтернативного сплайсинга — изображение увеличивается
Такая организация генов позволяет, например, осуществить процесс альтернативного сплайсинга, когда с одного гена могут быть синтезированы разные формы белка, за счет того, что в процессе сплайсинга экзоны могут сшиваться в разных последовательностях.
Сравнение строения генов прокариот и эукариот

Рис. 21. Отличия в строении генов прокариот и эукариот — изображение увеличивается
МУТАЦИИ И МУТАГЕНЕЗ
Мутацией называется стойкое изменение генотипа, то есть изменение нуклеотидной последовательности.
Процесс, который приводит к возникновению мутаций называется мутагенезом, а организм, все клетки которого несут одну и ту же мутацию — мутантом.
Мутационная теория была впервые сформулирована Гуго де Фризом в 1903 году. Современный ее вариант включает в себя следующие положения:
1. Мутации возникают внезапно, скачкообразно.
2. Мутации передаются из поколения в поколение.
3. Мутации могут быть полезными, вредными или нейтральными, доминантными или рецессивными.
4. Вероятность обнаружения мутаций зависит от числа исследованных особей.
5. Сходные мутации могут возникать повторно.
6. Мутации не направленны.
Мутации могут возникать под действием различных факторов. Различают мутации, возникшие под действием мутагенных воздействий: физических (например, ультрафиолета или радиации), химических (например, колхицина или активных форм кислорода) и биологических (например, вирусов). Также мутации могут быть вызваны ошибками репликации.
В зависимости от условий появления мутации подразделяют на спонтанные — то есть мутации, возникшие в нормальных условиях, и индуцированые — то есть мутации, которые возникли при особых условиях.
Мутации могут возникать не только в ядерной ДНК, но и, например, в ДНК митохондрий или пластид. Соответственно, мы можем выделять ядерные и цитоплазматические мутации.
В результате возникновения мутаций часто могут появляться новые аллели. Если мутантный аллель подавляет действие нормального, мутация называется доминантной. Если нормальный аллель подавляет мутантный, такая мутация называется рецессивной. Большинство мутаций, приводящих к возникновению новых аллелей являются рецессивными.
По эффекту выделяют мутации адаптивные, приводящие к повышению приспособленности организма к среде, нейтральные, не влияющие на выживаемость, вредные, понижающие приспособленность организмов к условиям среды и летальные, приводящие к смерти организма на ранних стадиях развития.
По последствиям выделяются мутации, приводящие к потери функции белка, мутации, приводящие к возникновению у белка новой функции, а также мутации, которые изменяют дозу гена, и, соответственно, дозу белка синтезируемого с него.
Мутация может возникнуть к любой клетке организма. Если мутация возникает в половой клетке, она называется герминативной (герминальной, или генеративной). Такие мутации не проявляются у того организма, у которого они появились, но приводят к появлению мутантов в потомстве и передаются по наследству, поэтому они важны для генетики и эволюции. Если мутация возникает в любой другой клетке, она называется соматической. Такая мутация может в той или иной степени проявляться у того организма, у которого она возникла, например, приводить к образованию раковых опухолей. Однако такая мутация не передается по наследству и не влияет на потомков.
Мутации могут затрагивать разные по размеру участки генома. Выделяют генные, хромосомные и геномные мутации.
Генные мутации
Мутации, которые возникают в масштабе меньшем, чем один ген, называются генными, или точечными (точковыми). Такие мутации приводят к изменению одного и нескольких нуклеотидов в последовательности. Среди генных мутаций выделяют замены, приводящие к замене одного нуклеотида на другой, делеции, приводящие к выпадению одного из нуклеотидов, инсерции, приводящие к добавлению лишнего нуклеотида в последовательность.

Рис. 23. Генные (точечные) мутации
По механизму воздействия на белок, генные мутации делят на: синонимичные, которые (в результате вырожденности генетического кода) не приводят к изменению аминокислотного состава белкового продукта, миссенс-мутации, которые приводят к замене одной аминокислоты на другую и могут влиять на структуру синтезируемого белка, хотя часто они оказываются незначительными, нонсенс-мутации, приводящие к замене кодирующего кодона на стоп-кодон, мутации, приводящие к нарушению сплайсинга:

Рис. 24. Схемы мутаций
Также по механизму воздействия на белок выделяют мутации, приводящие к сдвигу рамки считывания, например, инсерции и делеции. Такие мутации, как и нонсенс-мутации, хоть и возникают в одной точке гена, часто воздействуют на всю структуру белка, что может привести к полному изменению его структуры.
Рис. 25. Схема мутации, приводящей к сдвигу рамки считывания
Хромосомные мутации

Рис. 26. Хромосомные абберации
Хромосомными мутациями называются мутации, которые затрагивают отдельные гены в рамках одной хромосомы. Различают делеции, когда теряется один или несколько генов, дупликации, когда удваивается тот или иной ген или несколько генов, инверсии, когда участок хромосомы поворачивается на 180 градусов, транслокации, когда гены переходят с одной хромосомы на другую.

Рис. 27. Схемы хромосомных мутаций: делеции, дупликации, инверсии
|
|
|
|
Рис. 28. Транслокация |
Рис. 29. Хромосома до и после дупликации |
Геномные мутации
Наконец, геномные мутации затрагивают весь геном целиком, то есть меняется количество хромосом. Выделяют полиплоидии — увеличение плоидности клетки, и анеуплоидии, то есть изменение количества хромосом, например, трисомии (наличие у одной из хромосом дополнительного гомолога) и моносомии (отсутствие у хромосомы гомолога).
Видео по теме ДНК
РЕПЛИКАЦИЯ ДНК, КОДИРОВАНИЕ РНК, СИНТЕЗ БЕЛКА
(Если видео не отображается оно доступно по ссылке→)
См. дополнительно:
- Нуклеиновые кислоты (PDF)
- Общие сведения о секвенировании биополимеров
- Метагеномика и микробиом
- Бактериальный иммунитет и система CRISPR/Cas
- Трансляция белка на рисбосоме (общие сведения)
- Раскрыт секрет спиральной структуры ДНК (новое о ДНК)
- Антимутагенные свойства пробиотиков (в свете защиты ДНК)
- МикроРНК, микробиом кишечника и иммунитет
- Эпигенетика, короткоцепочечные жирные кислоты и врожденная иммунная память
- Замедление старения: роль питательных веществ и микробиоты в модуляции эпигенома (о метилировании ДНК)
Литература в помощь:
Будьте здоровы!
ССЫЛКИ К РАЗДЕЛУ О ПРЕПАРАТАХ ПРОБИОТИКАХ
- ПРОБИОТИКИ
- ПРОБИОТИКИ И ПРЕБИОТИКИ
- СИНБИОТИКИ
- ДОМАШНИЕ ЗАКВАСКИ
- КОНЦЕНТРАТ БИФИДОБАКТЕРИЙ ЖИДКИЙ
- ПРОПИОНИКС
- ЙОДПРОПИОНИКС
- СЕЛЕНПРОПИОНИКС
- БИФИКАРДИО
- ПРОБИОТИКИ С ПНЖК
- МИКРОЭЛЕМЕНТНЫЙ СОСТАВ
- БИФИДОБАКТЕРИИ
- ПРОПИОНОВОКИСЛЫЕ БАКТЕРИИ
- МИКРОБИОМ ЧЕЛОВЕКА
- МИКРОФЛОРА ЖКТ
- ДИСБИОЗ КИШЕЧНИКА
- МИКРОБИОМ и ВЗК
- МИКРОБИОМ И РАК
- МИКРОБИОМ, СЕРДЦЕ И СОСУДЫ
- МИКРОБИОМ И ПЕЧЕНЬ
- МИКРОБИОМ И ПОЧКИ
- МИКРОБИОМ И ЛЕГКИЕ
- МИКРОБИОМ И ПОДЖЕЛУДОЧНАЯ ЖЕЛЕЗА
- МИКРОБИОМ И ЩИТОВИДНАЯ ЖЕЛЕЗА
- МИКРОБИОМ И КОЖНЫЕ БОЛЕЗНИ
- МИКРОБИОМ И КОСТИ
- МИКРОБИОМ И ОЖИРЕНИЕ
- МИКРОБИОМ И САХАРНЫЙ ДИАБЕТ
- МИКРОБИОМ И ФУНКЦИИ МОЗГА
- АНТИОКСИДАНТНЫЕ СВОЙСТВА
- АНТИОКСИДАНТНЫЕ ФЕРМЕНТЫ
- АНТИМУТАГЕННАЯ АКТИВНОСТЬ
- МИКРОБИОМ и ИММУНИТЕТ
- МИКРОБИОМ И АУТОИММУННЫЕ БОЛЕЗНИ
- ПРОБИОТИКИ и ГРУДНЫЕ ДЕТИ
- ПРОБИОТИКИ, БЕРЕМЕННОСТЬ, РОДЫ
- ВИТАМИННЫЙ СИНТЕЗ
- АМИНОКИСЛОТНЫЙ СИНТЕЗ
- АНТИМИКРОБНЫЕ СВОЙСТВА
- КОРОТКОЦЕПОЧЕЧНЫЕ ЖИРНЫЕ КИСЛОТЫ
- СИНТЕЗ БАКТЕРИОЦИНОВ
- АЛИМЕНТАРНЫЕ ЗАБОЛЕВАНИЯ
- МИКРОБИОМ И ПРЕЦИЗИОННОЕ ПИТАНИЕ
- ФУНКЦИОНАЛЬНОЕ ПИТАНИЕ
- ПРОБИОТИКИ ДЛЯ СПОРТСМЕНОВ
- ПРОИЗВОДСТВО ПРОБИОТИКОВ
- ЗАКВАСКИ ДЛЯ ПИЩЕВОЙ ПРОМЫШЛЕННОСТИ
- НОВОСТИ
Статья на конкурс «био/мол/текст»: ДНК — двойная спираль? Не всегда. Отдельные островки наших молекул наследственности могут по ошибке принимать довольно экзотические формы. Например, сворачиваться в спирали из четырех полигуаниновых нитей — вопреки классическим принципам молекулярной биологии. Но действительно ли подобные аномалии возникают «по ошибке»? Или природа давно уже «оседлала» эту странность нуклеиновых кислот, поставив её себе на службу? Можно ли считать четверные G-спирали рабочими «деталями» сложнейшей машины геномной регуляции? И случайна ли их причастность к процессам старения и канцерогенеза?
В мистическом фильме Д. Аронофски «Фонтан» присутствует весьма интересный образ. Конкистадор, отправившийся по велению испанской королевы на поиски древа вечной жизни (далекими прототипами героев здесь, по-видимому, послужили первооткрыватель Флориды Понсе де Леон и король Испании Фердинанд), находит его в храме индейцев Майя в Южной Америке. Однако, вкусив млечного сока этого древа, герой понимает, что что-то пошло не так. Вместо того чтобы обрести бессмертие, он начинает прорастать цветущей травой и полностью становится субстратом для этой буйной, паразитической по сути, растительности. Иными словами, у человека и у природы могут быть разные, если можно так выразиться, представления о жизненной силе и долголетии. И, возможно, именно поэтому в поисках путей продления жизни, мы постоянно натыкаемся на опасность канцерогенеза. Будь это исследования в области стволовых клеток, попытки преодолеть так называемый предел Хейфлика с помощью фермента теломеразы либо иные способы борьбы с клеточным старением. Всякий раз геронтология и онкология идут «рука об руку», теснейшим образом переплетаясь. И одной из точек сопряжения этих двух областей можно считать удивительные структурные аномалии ДНК, носящие название G-квадруплексов…
ДНК не по канону
Мы привыкли думать о ДНК как о двойной спирали, в которой азотистые основания нуклеотидов на противоположных цепях однозначно соответствуют друг другу: аденин — тимину, гуанин — цитозину. Эта, безусловно, фундаментальная особенность нуклеиновых кислот лежит в основе механизмов наследственности. Именно благодаря ей становятся возможными удвоение и корректирование ДНК, а также реализация генетической информации в структуре РНК и белков.
Однако на деле наши молекулы наследственности оказываются куда гибче, подвижнее и причудливее, нежели это было некогда описано легендарными нобелевскими лауреатами Дж. Уотсоном и Ф. Криком. Например, подобно РНК, ДНК может формировать так называемые шпильки, которые на двойной спирали приобретают вид крестообразных структур (рис. 1). Эти «аномальные» образования принимают активное участие в регуляции работы с генетической информацией и задействованы как в копировании ДНК (репликации) [1], так и в переносе информации с ДНК на РНК (транскрипции) [2].
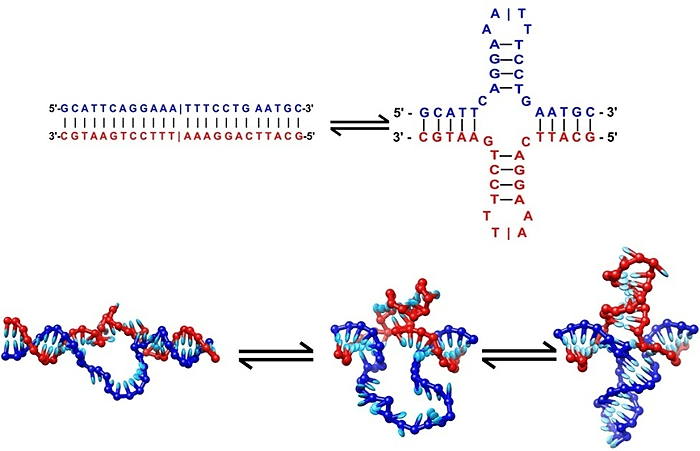
Рисунок 1. Формирование крестоообразных структур ДНК. Рисунок с сайта www.physoc.org.
Конечно, «крестами» и шпильками дело не ограничивается. К числу возможных конформаций ДНК относятся также тройные (рис. 2Б) и даже четверные спирали (рис. 2В), возникающие в результате неканонических связей между азотистыми основаниями [3]. В течение последнего десятилетия заметно возросло внимание к так называемым G-квадруплексам — структурам, представляющим собой спирали из четырех нитей ДНК или РНК, соединенных одними только гуанинами. Постепенно выясняется, что эти образования играют весьма важную роль в регуляции активности генов, в генетической изменчивости и в функционировании теломер [4].

Рисунок 2. Двойная (а), тройная (б) и четверная (в) спирали ДНК. Рисунок с сайта www.konan-fiber.jp.
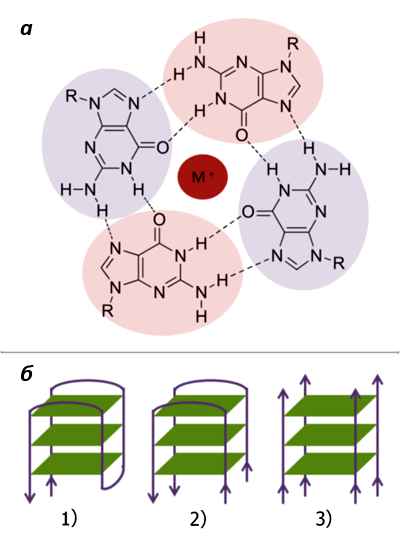
Рисунок 3. «G-ДНК». а. Строение G-тетрады; М+ — одновалентный катион. б. Формирование G-квадруплекса из 1) одной, 2) двух и 3) четырех нитей ДНК. Рисунок с сайта www.chem.cmu.edu.
Способность гуанина к самоассоциации была обнаружена еще в конце 19 века. И только в 1962 году удалось установить, что в растворах он образует агрегаты из четырех молекул (называемых G-квартетом, или G-тетрадой) [5]. Такие тетрады скрепляются между собой неканоническими (то есть, не предусмотренными в модели Уотсона — Крика) водородными связями, называемыми «хугстиновскими» — по фамилии их первооткрывателя [6]. При этом входящие в них гуанины располагаются в одной плоскости и нуждаются в стабилизации моновалентными катионами (например, K+ или Na+) (рис. 3а).
Содержащие гуанин нуклеиновые кислоты в растворе могут образовывать такие структуры из одной, двух или четырех различных нитей (рис. 3б). Однако стабильными они будут лишь в том случае, когда три и более G-тетрады сгруппируются в плотную «стопку», «подперев» друг друга межплоскостными стекинг-взаимодействиями и сформировав тем самым G-квадруплекс (G4-структуру). А для этого, в свою очередь, необходима «счастливая встреча» четырех полигуаниновых участков (GGGn), находящихся либо на одной, либо на разных молекулах ДНК или РНК [7].
Правда, на каждое правило есть свои исключения. Здесь можно привести в пример синдром ломкой X-хромосомы, возникающий вследствие экспансии многочисленных (более 200) повторов (CGG) в гене FMR1, необходимом для развития нервной системы. Матричная РНК такого мутантного гена способна формировать вполне стабильные четверные спирали даже из двухгуаниновых мотивов. То есть, в нашей «стопке» будет всего лишь две G-тетрады (рис. 4) [10].
«Значит, это кому-нибудь нужно?» © В. Маяковский
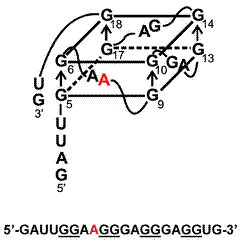
Рисунок 4. «Двуслойный» квадруплекс, формирующийся из четырех мотивов (GG) на РНК гена FMR1 при синдроме ломкой X-хромосомы. Рисунок с сайта www.computer.org.
Разумеется, то, что возможно в пробирке, далеко не всегда имеет место в живой клетке. Однако последние годы было убедительно показано, что G-квадруплексы действительно образуются in vivo в хромосомах живых организмов [11, 12]. И, кроме того, методами биоинформатики в геноме человека обнаружено порядка 376 тысяч районов, в которых потенциально могут возникать эти четырехспиральные образования. Интересно, что такие участки, как правило, находятся в составе регуляторных генетических элементов (промоторов, терминаторов, нетранслируемых последовательностей матричной РНК), теломер, рибосомных РНК и интронов, то есть последовательностей, не кодирующих структуру белка. Тогда как кодирующие фрагменты ДНК (экзоны) преимущественно «очищены» от подобных нуклеотидных мотивов [13, 14]. И неудивительно! Ведь, забегая вперед, нельзя не отметить, что такие гуаниновые «узелки» способны становиться очагами генетической нестабильности, провоцируя появление серьезных мутаций. Поэтому направленность естественного отбора в данном случае вполне понятна.
Однако почему-то всё тот же естественный отбор способствовал заметному скоплению потенциальных G4-структур в области генных промоторов, то есть, участков ДНК, определяющих производительность данного гена и позволяющих её регулировать [13]. Причем особенно ярко это выражено в группе онкогенов, повышенная активность которых необходима для возникновения и развития раковых опухолей. Тогда как, к примеру, встречаемость вероятных G-квадруплексов в промоторах генов — супрессоров раковых опухолей (призванных подавлять канцерогенез) значительно ниже, чем в среднем по геному [15, 16]. Более того, последовательности многих промоторов и интронов, особо обогащенных потенциальными G4-структурами, достаточно консервативны, то есть несут общие черты у целого ряда эукариотических организмов [17, 18].
Картину дополняет то обстоятельство, что живые системы не пожалели времени и сил на выработку специальных белков, способных связываться с G-квадруплексами, либо формируя и стабилизируя их, либо «разворачивая», «расплетая» или просто разрезая [19]. Бесполезным или «не существующим» в клетке структурам (каковыми их вплоть до недавнего времени считали многие исследователи) эволюция столько внимания не уделяет. Как отметил более 30 лет назад нобелевский лауреат Аарон Клуг: «Если G-квадруплексы так легко формируются in vitro, природа, должно быть, нашла путь применения их in vivo» [4]. С каждым годом мы получаем всё больше подтверждений этому прозорливому высказыванию.
Из двойной спирали — в четверную
Чтобы сформировать в ДНК такую крупную структуру, как G-квадруплекс, необходимо предварительно подвергнуть плавлению (разъединить) соответствующий участок классической двойной спирали. Длина таких участков может составлять несколько десятков нуклеотидов. К примеру, общая формула для поиска потенциальных G4-структур в геноме, использованная в работах исследователей из Кэмбриджского университета, выглядела как (G3+N1—7G3+N1—7G3+N1—7G3) — то есть, предполагала длину квадруплекса от 15 до 33 пар оснований [13] (хотя это далеко не единственный вариант [14]). Теоретически G-квадруплексы могут возникнуть практически в любом месте генома— при условии образования достаточно длинного однонитевого ДНК-фрагмента [16]. Другое дело, что по показателям стабильности они будут существенно проигрывать.
Наиболее благоприятные условия для высвобождения протяженных кусочков одноцепочечной ДНК создаются, прежде всего, во время репликации. И действительно: количество G4-структур в клетке заметно (приблизительно в пять раз) возрастает именно при удвоении хромосом [20]. В идеале, по завершении копирования наследственного материала все гуаниновые «узелки» (и старые, и новые) должны удаляться, поскольку в это время клетка буквально «причесывает» свою ДНК. Однако, как показывает практика, часть G-квадруплексов остается и присутствует в геноме на протяжении всего клеточного цикла [11, 12, 20]. Более того, по-видимому, сам процесс репликации нуждается в наличии G4-структур. В частности, они входят в состав ориджинов (геномных районов, с которых начинается репликация) позвоночных животных и, по одной из версий, определяют направление движения ДНК-полимеразы — «копировальной машины» ДНК [21, 22].
Образованию четверных гуаниновых спиралей могут способствовать не только репликация, но и транскрипция (считывание гена), а также репарация (починка) хромосом [23]. Кроме того, в ядре возможно спонтанное плавление двойной спирали ДНК, возникающее в результате тех или иных молекулярных эффектов [24, 25]. В конце концов, появление G4-структур может быть обусловлено целенаправленным воздействием на ДНК специальных белков — шаперонов, призванных формировать квадруплексы там, где это положено [19].
Куда легче дело обстоит с РНК, однонитевыми по своей природе молекулами. Исключительная гибкость и отсутствие «обременения» в виде второй комплементарной цепочки позволяет им свободно принимать самые разнообразные конформации. И не случайно потенциальные G4-спирали нашлись в нетранслируемых (то есть, входящих в состав матричной РНК, но структуру белка не кодирующих) областях более чем 3000 человеческих генов [26, 27]. Кроме того, в последние годы перспективным представляется исследование гибридных квадруплексов, состоящих из ДНК и РНК. Интересно, что для их формирования достаточно не четырех, а всего лишь двух полигуаниновых участков на ДНК (два других автоматически будут присутствовать на считываемой с гена РНК) [28].
На самом краю хромосомы
А теперь — всё внимание таким удивительным ядерным структурам, как теломеры [29]! Располагаясь на концах линейных хромосом ядерных организмов, они призваны защищать генетический материал от разрушения. Структуры всех эукариотических теломер удивительно консервативны: они представляют собой цепочку из многократно повторяющихся нуклеотидных мотивов. В человеческом геноме это мотив (TTAGGG) (обратим внимание читателя на тройной гуанин), воспроизведенный 1000–2000 раз [30]! Не правда ли, идеальная среда для возникновения многочисленных G-квадруплексов? Но и это еще не всё: на конце теломер человека и других теплокровных животных располагается довольно протяженный, 30–300 нуклеотидов, участок однонитевой ДНК со свободным 3’-концом [31]. Состоит он из всё тех же повторов (ТТАGGG). И уж здесь-то образованию гуаниновых узелков ничто не мешает.
Теломерные последовательности, будучи извлеченными из ядра или синтезированными, действительно массово формируют G-квадруплексы [4]. Однако доказательство их наличия в клетке требует куда более тонких подходов. С помощью меченых антител, специфически связывающихся с G4-структурами, было показано, что последние действительно образуются на концах хромосом (правда, не всех). По крайней мере, около 20% всех сигналов были зафиксированы именно там [20]. Возникают ли обнаруженные квадруплексы собственно в теломерах либо в близлежащих областях хромосом, по этим данным сказать трудно, поскольку разрешение метода сильно ограничено. Однако нам достоверно известно, что многие теломерные белки действительно взаимодействуют с G4-структурами (либо формируя их, либо разрушая), и это один из главных аргументов в пользу их полноценного участия в жизни теломер [19]. А уж о том, что эта «жизнь» чрезвычайно важна для нас в контексте вопроса о здоровье и долголетии, говорить не приходится.
Судя по всему, G-квадруплексы задействованы в формировании защитного колпачка, или кэпа, на конце теломеры. В клетках человека такое «кэпирование» осуществляется шелтерином — комплексом из шести белков. Он предохраняет линейную хромосому от излишнего внимания клеточных «контролёров», считающих, что конец цепочки ДНК — это повреждение, требующее соответствующих методов «лечения». А в области теломер такое «лечение» оборачивается катастрофическими последствиями: «сшивкой» разных хромосом и, в итоге, гибелью клетки либо запуском канцерогенеза [32].
С G4-структурами взаимодействуют такие шелтериновые белки, как TRF2 и POT1. При этом TRF2 связывает и стабилизирует квадруплексы на однонитевом 3’-фрагменте, тогда как POT1 обусловливает распад гуаниновых четверных спиралей [19]. Иными словами, квадруплексы при образовании «кэпа» нужны, но дозированно.
Предполагаются самые разнообразные варианты участия G4 ДНК в «кэпировании» хромосом [33]. G-квадруплекс может быть необходим для образования специфической теломерной структуры, именуемой T-петлей (теломерной петлей) (рис. 5, 6а). Он возникает либо при простом запетливании однонитевого 3’-фрагмента (рис. 6б), либо при его внедрении в двойную спираль теломерной ДНК (рис. 6в) [34]. Таким образом осуществляется маскировка свободного 3’-конца (в общем случае распознаваемого клеткой как сигнал тревоги) [33].
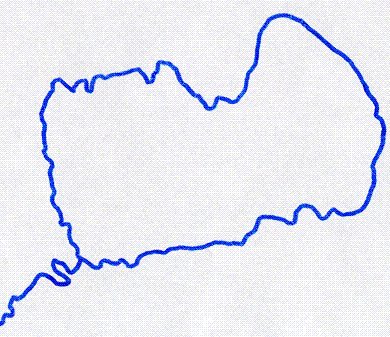
Рисунок 5. Т-петля на конце теломеры под электронным микроскопом. Фото с сайта web.pdx.edu.
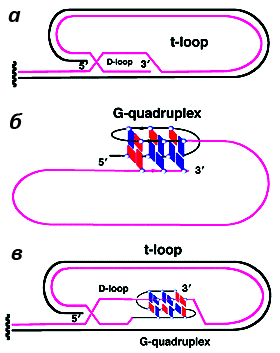
Рисунок 6. Различные виды Т-петель. а. T-петля без квадруплекса; б. T-петля с квадруплексом, замыкающим однонитевой 3’-фрагмент теломеры на себя; в. T-петля, образующая квадруплекс при внедрении однонитевого 3’-конца в двойную спираль теломерной ДНК. Рисунок с сайта pubs.rsc.org.
Помимо этого, обсуждаются альтернативные T-петле способы «кэпирования» хромосом. По одной из версий, одноцепочечная 3’-ДНК формирует плотный ряд G-квадруплексов, стабилизирующих однонитевой фрагмент и предохраняющих его от деградации особо ретивыми клеточными белками (рис. 7) [35]. Вероятно, подобный способ защиты теломеры используется в тех случаях, когда полноценного шелтерина на конце хромосомы не образуется [36].
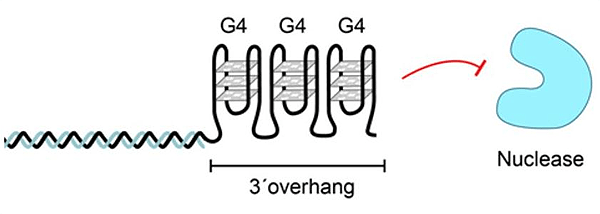
Рисунок 7. Защита теломеры с помощью серии G-квадруплексов, образующихся на однонитевом 3’-конце. Рисунок из [4].
Не исключено, что в клетках человека в той или иной мере реализуются оба способа «обороны» теломеры с участием G4-образований. Иное дело, что такая «оборона» может оборачиваться для нас рядом неприятных моментов.
Точка обратного отсчета
Согласно наиболее популярной точке зрения, именно в теломерах спрятан ключ (по крайней мере, один из главных таких ключей) к пониманию процесса старения и ограниченной продолжительности нашей жизни. Линейные хромосомы, в отличие от циклических, коими могут похвастаться большинство доядерных организмов, как правило, не способны удваиваться вечно. Каждый цикл репликации, в силу особенностей организации этого ферментативного механизма, приводит к укорочению одной из дочерних нитей ДНК на 50–100 нуклеотидов. В итоге, после 50–60 раундов копирования наследственного материала потери в длине теломеры становятся неприемлемыми, поэтому клетка запускает процесс своего старения (сенесценции) либо гибнет [30, 37]. Правда, всем известные стволовые клетки являются ярким исключением из этого общего правила [38, 39]. (А еще — клетки растений, меняющие длину теломер в зависимости от времени года [40].)
Исследуя механизмы старения человека, невозможно обойти вниманием такое наследственное заболевание как синдром Вернера. Проявляется оно в ускоренном старении организма и сопровождается всеми сопутствующими эффектами: снижением иммунитета, повышением риска раковых заболеваний, многочисленными физиологическими нарушениями, характерными для людей преклонного возраста. Причиной этого синдрома оказалось нарушение структуры белка — геликазы WRN, основной функцией которого является расплетание теломерных квадруплексов. В результате во время репликации синтез дочерней цепи ДНК тормозится и обрывается в месте образования G4-структуры. Это приводит к галопирующему укорочению теломер и, соответственно, раннему запуску клеточного старения [41]. Аналогичного эффекта в культурах клеток можно добиться искусственным путем, повышая стойкость квадруплексов за счет их связывания со специальными химическими веществами [42].
Можно спорить по поводу применимости модели наследственного заболевания в деле расшифровки механизмов нормального, не патологичного старения. Однако совершенно не исключено, что не расплетенные вовремя теломерные квадруплексы задействованы и в этом случае. Вспомним, к примеру, как долгое время считалось, что синдром прогерии Хатчинсона-Гилфорда, связанный с преждевременным старением у детей, не имеет отношения к старению как таковому. В дальнейшем же было показано, что вызывающий эту болезнь прогерин образуется и в коже обычных пожилых людей [43].
Помимо этого, не секрет, что одной из вероятных причин старения является накопление в геноме повреждений, вызванных окислением свободными радикалами [44]. Причем существенная часть из них концентрируется именно на теломерах [45]. Возможных объяснений такому предпочтительному поражению кончиков хромосом довольно много. По-видимому, в этом виновны и сниженная (силами шелтериновых белков) активность ферментов «ремонта» ДНК, и особенности нуклеотидной последовательности теломерной ДНК. И здесь опять имеет смысл вспомнить про наши G-квадруплексы! Ведь они, помимо всего прочего, оказываются особенно чувствительными к окислительным повреждениям [46].
Смертельно опасная «вечная» жизнь
Говоря о теломерных G4-структурах, нельзя обойти вниманием тот интригующий факт, что, располагаясь на концах 3’-выступа, они блокируют работу теломеразы. А ведь это тот самый широко популяризованный фермент, на который возлагалось столько надежд, и за изучение которого в 2009 году была вручена Нобелевская премия по физиологии и медицине! Теломераза наращивает 3’-концы теломер и таким образом нивелирует их естественное укорочение. Иными словами — продлевает активную жизнь хромосомам и, соответственно, клетке [29]. Однако G-квадруплексы не просто мешают теломеразе делать свое дело — помимо этого, они, располагаясь в области промотора гена TERT, подавляют ее синтез в клетке [47]. И, конечно, этот эффект можно было бы счесть за негативный, если бы не один весьма печальный факт. Дело в том, что избыточная активность теломеразы создает весьма благоприятную среду для перерождения нормальных клеток в раковые. От работы этого фермента сильно зависят порядка 90% всех злокачественных опухолей, ведь он позволяет перерожденным клеткам делиться бесконечно долго [47].
Блокирование работы теломеразы составляет одну из функций гена BRCA1. BRCA1 — широко известный супрессор раковых опухолей, то есть ген — «борец» с канцерогенезом. Он является важным компонентом системы исправления ошибок в ДНК и контролирует активность целого ряда генов, в том числе задействованных в образовании опухолей [48]. Поэтому мутации в BRCA1 зачастую приводят к развитию рака груди и яичников [49], а в России они — в «лидерах» генетических причин возникновения онкозаболеваний [50]. Интересно, что подавление теломеразы этим (весьма немаловажным, как мы убедились) белком осуществляется не только за счет регуляции активности кодирующего её гена TERT [51], но и за счет прямого вмешательства в её работу на теломерах. В частности, предполагают, что BRCA1 связывается с теломерными квадруплексами и стабилизирует их, в результате чего наш «фермент молодости» остаётся не у дел [19].
Как было отмечено выше, TERT — отнюдь не единственный регулируемый G-квадруплексами ген, благоприятствующий развитию злокачественных опухолей [15]. Помимо него в этом ряду фигурируют c-MYC, c-KIT, BLC2, VEGF, HIF-1a и целый ряд других известных онкогенов [52]. Разумеется, такая закономерность не могла не привлечь внимания ученых, занятых поиском средств против рака. Сегодня существует целый ряд работ, посвященных влиянию тех или иных квадруплекс-связывающих веществ на рост и развитие раковых клеток. И, по результатам этих исследований, искусственное укрепление G-квадруплексов представляется чрезвычайно перспективным путем лечения онкологических заболеваний [47, 52].
По лезвию бритвы
Как гласит известная легенда, Будда Шакьямуни обрел просветление, услышав фразу учителя музыки, адресованную его ученику: «Если ты ослабишь струну, музыка не зазвучит, а если ты перетянешь её, она порвется». И этот принцип в полной мере относится ко множеству, если не ко всем биологическим процессам.
Те же самые G4-структуры, которые столь перспективны в рамках лечения раковых опухолей, могут сами по себе становиться провокаторами канцерогенных заболеваний. Вспомним вышеупомянутый синдром преждевременного старения Вернера. Возникает он на фоне недееспособности геликазы WRN, в норме «развязывающей» гуаниновые «узелки» на теломерах. В наших клетках существует целый спектр таких белков, призванных тем или иным способом удалять G-квадруплексы, дабы освободить дорогу ферментам репликации. Если же этого не происходит, то копирование ДНК стопорится в месте образования G4-структуры. В результате запускается целый ряд процессов, приводящих к разрывам ДНК, хромосомным перестройкам и мутациям наследственного материала [36, 41].
Так, например, при повреждении гена специализированной на G-квадруплексах геликазы BLM возникает синдром Блума. Проявляется он в низкорослости и ранней предрасположенности к целому ряду онкозаболеваний. Нарушение функции геликазы FANJ приводит к развитию анемии Фалькони, сопровождающейся повышенной хрупкостью хромосом и склонностью к лейкозам. Аналогичным образом мутации в гене геликазы RETL1 увеличивают чувствительность организма к некоторым видам рака. Этот список можно было бы продолжить целым рядом наследственных заболеваний, провоцирующих канцерогенез. И все они связаны с генетической и теломерной нестабильностью, вызванной «стойкими» ДНК- и РНК-квадруплексами [41].
Иными словами, чтобы «музыка», исполняемая оркестром генных и теломерных G-квадруплексов, звучала, необходима тонкая настройка их жесткости. По-видимому, мы имеем дело с чрезвычайно широкой и сложной сетью структурных ДНК-аномалий. Возникающих как спонтанно, так и при помощи специальных белков. Чувствительных как к молекулярному окружению, так и к физико-химическим условиям среды. Способных как предотвращать раковые заболевания, так и провоцировать их. Принимающих участие как в защите теломер, так и в процессах клеточного старения.
- Pearson C.E., Zorbas H., Price G.B., Zannis-Hadjopoulos M. (1996). Inverted repeats, stem-loops, and cruciforms: significance for initiation of DNA replication. J. Cell Biochem. 63, 1–22;
- Wadkins R.M. (2000). Targeting DNA secondary structures. Curr. Med. Chem. 7, 1–15;
- Франк-Каменецкий М. (2015). Необычные формы ДНК. Портал «ПостНаука»;
- Rhodes D. and Lipps H.J. (2015). G-quadruplexes and their regulatory roles in biology. Nucl. Acids Res. 43, 8627–8637;
- Gellert M., Lipsett M.N., Davies D.R. (1962). Helix formation by guanylic acid. Proc. Natl. Acad. Sci. USA. 48, 2013–2018;
- Hoogsteen K. (1963). The crystal and molecular structure of a hydrogen-bonded complex between 1-methylthymine and 9-methyladenine. Acta Cryst. 16, 907–916;
- Burge S., Parkinson G.N., Hazel P., Todd A.K, Neidle S. (2006). Quadruplex DNA: sequence, topology and structure. Nucl. Acids Res. 34, 5402–5415;
- Kotlyar A.B., Borovok N., Molotsky T., Cohen H., Shapir E., Porath D. (2005). Long, monomolecular guanine-based nanowires. Adv. Mat. 17, 1901–1905;
- Ngo V.A., Felice R.D., Haas S. (2014). Is the G-quadruplex an effective nanoconductor for ions? J. Phys. Chem. B. 118, 864–872;
- Kettania A., Kumara A.R., Patela D.J. (2014). Solution structure of a DNA quadruplex containing the fragile X syndrome triplet repeat. J. Mol. Biol. 254, 638–656;
- Lam E.Y., Beraldi D., Tannahill D., Balasubramanian S. (2013). G-quadruplex structures are stable and detectable in human genomic DNA. Nat. Commun. 4, 1796–1780;
- Rodriguez R., Miller K.M., Forment J.V., Bradshaw C.R., Nikan M., Britton S. et al. (2012). Small-molecule-induced DNA damage identifies alternative DNA structures in human genes. Nat. Chem. Biol. 8, 301–310;
- Huppert J.L. and Balasubramanian S. (2005). Prevalence of quadruplexes in the human genome. Nucl. Acids Res. 33, 2908–2916;
- Todd A.K., Johnston M., Neidle S. (2005). Highly prevalent putative quadruplex sequence motifs in human DNA. Nucl. Acids Res. 33, 2901–2907;
- Eddy J. and Maizels N. (2006). Gene function correlates with potential for G4 DNA formation in the human genome. Nucl. Acids Res. 34, 3887–3896;
- Maizels N. and Gray L.T. (2013). The G4 Genome. PLoS Genet. 9, e1003468;
- Fernando H., Reszka A.P., Huppert J., Ladame S., Rankin S., Venkitaraman A.R. et al. (2006). A conserved quadruplex motif located in a transcription activation site of the human c-kit oncogene. Biochem. 45, 7854–7860;
- Eddy J. and Maizels N. (2008). Conserved elements with potential to form polymorphic G-quadruplex structures in the first intron of human genes. Nucl. Acids Res. 36, 1321–1333;
- Brazda V., Haronikova L., Liao J.C., Fojta M. (2014). DNA and RNA quadruplex-binding proteins. Int. J. Mol. Sci. 15, 17493–17517;
- Biffi G., Tannahill D., McCafferty J., Balasubramanian S. (2013). Quantitative visualization of DNA G-quadruplex structures in human cells. Nat. Chem. 5, 182–186;
- Leonard A.C. and Mechali M. (2013). DNA replication origins. Cold Spring Harb. Perspect. Biol. 5, a010116;
- Valton A.L., Hassan-Zadeh V., Lema I., Boggetto N., Alberti P., Saintome C. et al. (2014). G4 motifs affect origin positioning and efficiency in two vertebrate replicators. EMBO J. 33, 732–746;
- Li W., Wu P., Ohmichi T., Sugimoto N. (2002). Characterization and thermodynamic properties of quadruplex/duplex competition. FEBS Lett. 526, 77–81;
- Miyoshi D., Karimata H., Sugimoto N. (2006). Hydration regulates thermodynamics of G-quadruplex formation under molecular crowding conditions. J. Am. Chem. Soc. 128, 7957–7963;
- Nikolova E.N., Kim E., Wise A.A., O’Brien P.J., Andricioaei I., Al-Hashimi H.M. (2011). Transient Hoogsteen base pairs in canonical duplex DNA. Nature. 470, 498–502;
- Bugaut A. and Balasubramanian S. (2012). 5′-UTR RNA G-quadruplexes: translation regulation and targeting. Nucl. Acids Res. 40, 4727–4741;
- Beaudoin J.D. and Perreault J.P. (2013). Exploring mRNA 3′-UTR G-quadruplexes: evidence of roles in both alternative polyadenylation and mRNA shortening. Nucl. Acids Res. 41, 5898–5911;
- Xiao S., Zhang J.Y., Zheng K.W., Hao Y.H., Tan Z. (2013). Bioinformatic analysis reveals an evolutional selection for DNA:RNA hybrid G-quadruplex structures as putative transcription regulatory elements in warm-blooded animals. Nucl. Acids Res. 41, 10379–10390;
- «Нестареющая» Нобелевская премия: в 2009 году отмечены работы по теломерам и теломеразе;
- Старение — плата за подавление раковых опухолей?;
- Makarov V.L., Hirose Y., Langmore J.P. (1997). Long G tails at both ends of human chromosomes suggest a C strand degradation mechanism for telomere shortening. Cell. 88, 657–666;
- Sfeir A. and Lange T. (2012). Removal of shelterin reveals the telomere end-protection problem. Science. 336, 593–597;
- Galati A., Micheli E., Cacchione S. (2013). Chromatin Structure in Telomere Dynamics. Front. Oncol. 3, 46;
- Табу в науке: лаборатория, задававшая неправильные вопросы;
- Bochman M.L., Paeschke K., Zakian V.A. (2012). DNA secondary structures: stability and function of G-quadruplex structures. Nat. Rev. Genet. 13, 770–780;
- Brosh R.M. Jr. (2011). Put on your thinking cap: G-quadruplexes, helicases, and telomeres. Aging (Albany NY). 3, 332–335;
- Levy M.Z., Allsopp R.Z., Futcher A.B., Greider C.W., Harley C.B. (1992). Telomere end-replication problem and cell aging. J. Mol. Biol. 225, 951–960;
- Была клетка простая, стала стволовая;
- Ствол и ветки: стволовые клетки;
- Длина теломер и времена года;
- Brosh R.M. Jr. (2013). DNA helicases involved in DNA repair and their roles in cancer. Nat. Rev. Cancer. 13, 542–558;
- Incles C.M., Schultes C.M., Kempski H., Koehler H., Kelland L.R., Neidle S. (2004). A G-quadruplex telomere targeting agent produces p16-associated senescence and chromosomal fusions in human prostate cancer cells. Mol. Cancer Ther. 3, 1201;
- Burtner C.R. and Kennedy B.K. (2010). Progeria syndromes and ageing: what is the connection? Nat. Rev. Mol. Cell Biol. 11, 567–578;
- Wickens A.P. (2001). Ageing and the free radical theory. Respir. Physiol. 128, 379–391;
- Sun L., Tan R., Xu J., LaFace J., Gao Y., Xiao Y. et al. (2015). Targeted DNA damage at individual telomeres disrupts their integrity and triggers cell death. Nucl. Acids Res. 43, 6334–6347;
- Clark D.W., Phang T., Edwards M.G., Geraci M.W., Gillespie M.N. (2012). Promoter G-quadruplex sequences are targets for base oxidation and strand cleavage during hypoxia-induced transcription. Free Radic. Biol. Med. 53, 51–59;
- Patel D.J., Phan A.T., Kuryavyi V. (2007). Human telomere, oncogenic promoter and 5′-UTR G-quadruplexes: diverse higher order DNA and RNA targets for cancer therapeutics. Nucl. Acids Res. 35, 7429–7455;
- Rosen E.M. (2013). BRCA1 in the DNA damage response and at telomeres. Front. Genet. 4, 85;
- Рак молочной железы с семейной историей;
- Любченко Л.Н. Наследственный рак молочной железы и/или яичников: ДНК-диагностика, индивидуальный прогноз, лечение и профилактика: дис. … д-ра мед. наук. — Москва, 2009;
- Xiong J., Fan S., Meng Q., Schramm L., Wang C., Bouzahza B. et al. (2003). BRCA1 inhibition of telomerase activity in cultured cells. Mol. Cell Biol. 23, 8668–8690;
- Balasubramanian S., Hurley L.H., Neidle S. (2011). Targeting G-quadruplexes in gene promoters: a novel anticancer strategy? Nat. Rev. Drug Discov. 10, 261–275..
Двойная спираль ДНК
Дезоксирибонуклеи́новая кислота́ (ДНК) — один из двух типов нуклеиновых кислот, обеспечивающих хранение, передачу из поколения в поколение и реализацию генетической программы развития и функционирования живых организмов. Основная роль ДНК в клетках — долговременное хранение информации о структуре РНК и белков.
В клетках эукариот (например, животных или растений) ДНК находится в ядре клетки в составе хромосом, а также в некоторых клеточных органоидах (митохондриях и пластидах). В клетках прокариотических организмов (бактерий и архей) кольцевая или линейная молекула ДНК, так называемый нуклеоид, прикреплена изнутри к клеточной мембране. У них и у низших эукариот (например, дрожжей) встречаются также небольшие автономные, преимущественно кольцевые молекулы ДНК, называемые плазмидами. Кроме того, одно- или двухцепочечные молекулы ДНК могут образовывать геном ДНК-содержащих вирусов.
С химической точки зрения, ДНК — это длинная полимерная молекула, состоящая из повторяющихся блоков, нуклеотидов. Каждый нуклеотид состоит из азотистого основания, сахара (дезоксирибозы) и фосфатной группы. Связи между нуклеотидами в цепи образуются за счёт дезоксирибозы и фосфатной группы. В подавляющем большинстве случаев (кроме некоторых вирусов, содержащих одноцепочечную ДНК) макромолекула ДНК состоит из двух цепей, ориентированных азотистыми основаниями друг к другу. Эта двухцепочечная молекула спирализована. В целом структура молекулы ДНК получила название «двойной спирали».
В ДНК встречается четыре вида азотистых оснований (аденин, гуанин, тимин и цитозин). Азотистые основания одной из цепей соединены с азотистыми основаниями другой цепи водородными связями согласно принципу комплементарности: аденин соединяется только с тимином, гуанин — только с цитозином. Последовательность нуклеотидов позволяет «кодировать» информацию о различных типах РНК, наиболее важными из которых являются информационные, или матричные (мРНК), рибосомальные (рРНК) и транспортные (тРНК). Все эти типы РНК синтезируются на матрице ДНК за счёт копирования последовательности ДНК в последовательность РНК, синтезируемой в процессе транскрипции и принимают участие в биосинтезе белков (процессе трансляции). Помимо кодирующих последовательностей, ДНК клеток содержит последовательности, выполняющие регуляторные и структурные функции. Кроме того, в геноме эукариот часто встречаются участки, принадлежащие «генетическим паразитам», например, транспозонам.
Расшифровка структуры ДНК (1953 г.) стала одним из поворотных моментов в истории биологии. За выдающийся вклад в это открытие Фрэнсису Крику, Джеймсу Уотсону, Морису Уилкинсу была присуждена Нобелевская премия по физиологии и медицине 1962 г.
Содержание
- 1 История изучения
- 2 Структура молекулы
- 2.1 Нуклеотиды
- 2.2 Двойная спираль
- 2.3 Образование связей между основаниями
- 2.4 Химические модификации оснований
- 2.5 Повреждение ДНК
- 2.6 Суперскрученность
- 2.7 Структуры на концах хромосом
- 3 Биологические функции
- 3.1 Структура генома
- 3.2 Последовательности генома, не кодирующие белок
- 3.3 Транскрипция и трансляция
- 3.4 Репликация
- 4 Взаимодействие с белками
- 4.1 Структурные и регуляторные белки
- 4.2 Ферменты, модифицирующие ДНК
- 4.2.1 Топоизомеразы и хеликазы
- 4.2.2 Нуклеазы и лигазы
- 4.2.3 Полимеразы
- 5 Генетическая рекомбинация
- 6 Эволюция метаболизма, основанного на ДНК
- 7 См. также
- 8 Ссылки
- 9 Рекомендуемая литература
- 10 Литература
История изучения


ДНК была открыта Иоганном Фридрихом Мишером в 1869 году. Вначале новое вещество получило название нуклеин, а позже, когда Мишер определил, что это вещество обладает кислотными свойствами, вещество получило название нуклеиновая кислота [1]. Биологическая функция новооткрытого вещества была неясна, и долгое время ДНК считалась запасником фосфора в организме. Более того, даже в начале XX века многие биологи считали, что ДНК не имеет никакого отношения к передаче информации, поскольку строение молекулы, по их мнению, было слишком однообразным и не могло содержать закодированную информацию.
Постепенно было доказано, что именно ДНК, а не белки, как считалось раньше, является носителем генетической информации. Одно из первых решающих доказательств принесли эксперименты О. Эвери, Колина Мак-Леода и Маклин Мак-Карти (1944 г.) по трансформации бактерий. Им удалось показать, что за так называемую трансформацию (приобретение болезнетворных свойств безвредной культурой в результате добавления в неё мёртвых болезнетворных бактерий) отвечают выделенные из пневмококков ДНК. Эксперимент американских учёных Алфреда Херши и Марты Чейз (Эксперимент Херши—Чейз, 1952 г.) с помеченными радиоактивными изотопами белками и ДНК бактериофагов показали, что в заражённую клетку передаётся только нуклеиновая кислота фага, а новое поколение фага содержит такие же белки и нуклеиновую кислоту, как исходный фаг[2].
Вплоть до 50-х годов XX века точное строение ДНК, как и способ передачи наследственной информации, оставалось неизвестным. Хотя и было доподлинно известно, что ДНК состоит из нескольких цепочек, состоящих из нуклеотидов, никто не знал точно, сколько этих цепочек и как они соединены.
Структура двойной спирали ДНК была предложена Френсисом Криком и Джеймсом Уотсоном в 1953 году на основании рентгеноструктурных данных, полученных Морисом Уилкинсом и Розалинд Франклин, и «правил Чаргаффа», согласно которым в каждой молекуле ДНК соблюдаются строгие соотношения, связывающие между собой количество азотистых оснований разных типов[3]. Позже предложенная Уотсоном и Криком модель строения ДНК была доказана, а их работа отмечена Нобелевской премией по физиологии и медицине 1962 г. Среди лауреатов не было скончавшейся к тому времени Розалинды Франклин, так как премия не присуждается посмертно[4].
Структура молекулы
Нуклеотиды
Структуры оснований, наиболее часто встречающихся в составе ДНК
Дезоксирибонуклеиновая кислота (ДНК) представляет собой биополимер (полианион), мономером которого является нуклеотид[5][6].
Каждый нуклеотид состоит из остатка фосфорной кислоты присоединённого по 5′-положению к сахару дезоксирибозе, к которому также через гликозидную связь (C—N) по 1′-положению присоединено одно из четырёх азотистых оснований. Именно наличие характерного сахара и составляет одно из главных различий между ДНК и РНК, зафиксированное в названиях этих нуклеиновых кислот (в состав РНК входит сахар рибоза)[7]. Пример нуклеотида — аденозинмонофосфат — где основание, присоединённое к фосфату и рибозе, это аденин, показан на рисунке.
Исходя из структуры молекул, основания, входящие в состав нуклеотидов, разделяют на две группы: пурины (аденин [A] и гуанин [G]) образованы соединёнными пяти- и шестичленным гетероциклами; пиримидины (цитозин [C] и тимин [T]) — шестичленным гетероциклом[8].
В виде исключения, например, у бактериофага PBS1, в ДНК встречается пятый тип оснований — урацил ([U]), пиримидиновое основание, отличающееся от тимина отсуствием метильной группы на кольце, обычно заменяющее тимин в РНК[9].
Следует отметить, что тимин и урацил не так строго приурочены к ДНК и РНК соответственно, как это считалось ранее. Так, после синтеза некоторых молекул РНК значительное число урацилов в этих молекулах метилируется с помощью специальных ферментов, превращаясь в тимин. Это происходит в транспортных и рибосомальных РНК[10].

В зависимости от концентрации ионов и нуклеотидного состава молекулы, двойная спираль ДНК в живых организмах существует в разных формах. На рисунке (слева направо) представлены A, B и Z формы
Двойная спираль
Полимер ДНК обладает довольно сложной структурой. Нуклеотиды соединены между собой ковалентно в длинные полинуклеотидные цепи. Эти цепи в подавляющем большинстве случаев (кроме некоторых вирусов, обладающих одноцепочечными ДНК-геномами), в свою очередь, попарно объединяются при помощи водородных связей в структуру, получившую название двойной спирали [3][7]. Остов каждой из цепей состоит из чередующихся фосфатов и сахаров[11]. Фосфатные группы формируют фосфодиэфирные связи между третьим и пятым атомами углерода соседних молекул дезоксирибозы, в результате взаимодействия между 3′-гидроксильной (3′ —ОН) группой одной молекулы дезоксирибозы и 5′-фосфатной группой (5′ —РО3) другой. Асимметричные концы цепи ДНК называются 3′ (три прим) и 5′ (пять прим). Полярность цепи играет важную роль при синтезе ДНК (удлинение цепи возможно только путём присоединения новых нуклеотидов к свободному 3′ концу).
Как уже было сказано выше, у подавляющего большинства живых организмов ДНК состоит не из одной, а из двух полинуклеотидных цепей. Эти две длинные цепи закручены одна вокруг другой в виде двойной спирали, стабилизированной водородными связями, образующимися между обращёнными друг к другу азотистыми основаниями входящих в неё цепей. В природе эта спираль, чаще всего, правозакрученная. Направления от 3′ конца к 5′ концу в двух цепях, из которых состоит молекула ДНК, противоположны (цепи «антипараллельны» друг другу).
Ширина двойной спирали составляет от 22 до 24 Å (ангстрем), или 2,2 — 2,4 нанометра, длина каждого нуклеотида 3,3 Å (0,33 нанометра)[12]. Подобно тому, как в винтовой лестнице сбоку можно увидеть ступеньки, на двойной спирали ДНК в промежутках между фосфатным остовом молекулы можно видеть ребра оснований, кольца которых расположены в плоскости, перпендикулярной по отношению к продольной оси макромолекулы.
В двойной спирали различают малую (12 Å) и большую (22 Å) бороздки[13]. Белки, например, факторы транскрипции, которые присоединяются к определённым последовательностям в двухцепочечной ДНК, обычно взаимодействуют с краями оснований в большой бороздке, где те более доступны.[14].
Образование связей между основаниями
Каждое основание на одной из цепей связывается с одним определённым основанием на второй цепи. Такое специфическое связывание называется комплементарным. Пурины комплементарны пиримидинам (то есть, способны к образованию водородных связей с ними): аденин образует связи только с тимином, а цитозин — с гуанином. В двойной спирали цепочки также связаны с помощью гидрофобных связей и стэкинга, которые не зависят от последовательности оснований ДНК[15].
Комплементарность двойной спирали означает, что информация, содержащаяся в одной цепи, содержится и в другой цепи. Обратимость и специфичность взаимодействий между комплементарными парами оснований важна для репликации ДНК и всех остальных функций ДНК в живых организмах.
Так как водородные связи нековалентны, они легко разрываются и восстанавливаются. Цепочки двойной спирали могут расходиться как замок-молния под действием ферментов (хеликазы) или при высокой температуре[16]. Разные пары оснований образуют разное количество водородных связей. АТ связаны двумя, ГЦ — тремя водородными связями, поэтому на разрыв ГЦ требуется больше энергии. Процент ГЦ пар и длина молекулы ДНК определяют количество энергии, необходимой для диссоциации цепей: длинные молекулы ДНК с большим содержанием ГЦ более тугоплавки[17].
Части молекул ДНК, которые из-за их функций должны быть легко разделяемы, например ТАТА последовательность в бактериальных промоторах, обычно содержат большое количество А и Т.

Интеркалированное химическое соединение, которое находится в середине спирали -бензопирен, основной мутаген табачного дыма[18]
Химические модификации оснований
Структура хроматина влияет на транскрипцию генов: участки гетерохроматина (отсутствие или низкий уровень транскрипции генов) коррелирует с метилированием цитозина. Например, метилирование цитозина с образованием 5-метилцитозина важно для инактивации Х-хромосомы[19]. Средний уровень метилирования отличается у разных организмов, так у нематоды Caenorhabditis elegans метилирование цитозина не наблюдается, а у позвоночных обнаружен высокий уровень метилирования — до 1% [20].
Несмотря на биологическую роль, 5-метилцитозин может спонтанно утрачивать аминную группу (деаминироваться), превращаясь в тимин, поэтому метилированные цитозины являются источником повышенного числа мутаций[21]. Другие модификации оснований включают метилирование аденина у бактерий и гликозилирование урацила с образованием «J-основания» в кинетопластах[22].
Повреждение ДНК
ДНК может повреждаться разнообразными мутагенами, к которым относятся окисляющие и алкилирующие вещества, а также высокоэнергетическая электромагнитная радиация — ультрафиолетовое и рентгеновское излучение. Тип повреждения ДНК зависит от типа мутагена. Например, ультрафиолет повреждает ДНК путём образования в ней димеров тимина, которые образуются при образовании ковалентных связей между соседними основаниями[23]. Оксиданты, такие как свободные радикалы или перекись водорода приводят к нескольким типам повреждения ДНК, включая модификации оснований, в особенности гуанозина, а также двуцепочечные разрывы в ДНК [24]. По некоторым оценкам в каждой клетке человека окисляющими соединениями ежедневно повреждается порядка 500 оснований[25][26]. Среди разных типов повреждений наиболее опасные — это двуцепочечные разрывы, потому что они трудно репарируются и могут привести к потерям участков хромосом (делециям) и транслокациям.
Многие молекулы мутагенов вставляются (интеркалируют) между двумя соседними парами оснований. Большинство этих соединений, например, этидий, дауномицин, доксорубицин и талидомид имеют ароматическую структуру. Для того, чтобы интеркалирующее соединение могло поместиться между основаниями, они должны разойтись, расплетая и нарушая структуру двойной спирали. Эти изменения в структуре ДНК мешают транскрипции и репликации, вызывая мутации. Поэтому интеркалирующие соединения часто являются канцерогенами, наиболее известные из которых — бензопирен, акридины, афлатоксин и бромистый этидий[27][28][29]. Несмотря на эти негативные свойства, в силу их способности подавлять транскрипцию и репликацию ДНК, интеркалирующие соединения используются в химиотерапии для подавления быстро растущих клеток рака[30].
Суперскрученность
Если взяться за концы верёвки и начать скручивать их в разные стороны, она становится короче и на верёвке образуются «супервитки». Так же может быть суперскручена и ДНК. В обычном состоянии цепочка ДНК делает один оборот на каждые 10,4 основания, но в суперскрученном состоянии спираль может быть свёрнута туже или расплетена[31]. Выделяют два типа суперскручивания: положительное — в направлении нормальных витков, при котором основания расположены ближе друг к другу; и отрицательное — в противоположном направлении. В природе молекулы ДНК обычно находятся в отрицательном суперскручивании, которое вносится ферментами — топоизомеразами[32]. Эти ферменты удаляют дополнительное скручивание, возникающее в ДНК в результате транскрипции и репликации[33].
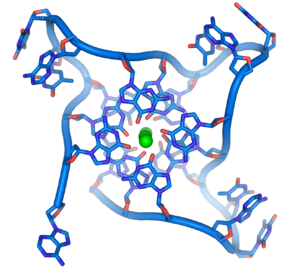
Структура теломер. Зелёным цветом показан ион металла, хелатированный в центре структуры[34]
Структуры на концах хромосом
На концах линейных хромосом находятся специализированные структуры ДНК, называемые теломерами. Основная функция этих участков — поддержание целостности концов хромосом[35]. Теломеры также защищают концы ДНК от деградации экзонуклеазами и предотвращают активацию системы репарации[36]. Поскольку обычные ДНК-полимеразы не могут реплицировать 3′ концы хромосом, это делает специальный фермент — теломераза.
В клетках человека теломеры обычно представлены одноцепочечной ДНК и состоят из несколько тысяч повторяющихся единиц последовательности ТТАГГГ[37]. Эти последовательности с высоким содержанием гуанина стабилизируют концы хромосом, формируя очень необычные структуры, называемые G-квадруплексами и состоящие из четырёх, а не двух взаимодействующих оснований. Четыре гуаниновых основания, все атомы которых находятся в одной плоскости, образуют пластинку, стабилизированную водородными связями между основаниями и хелатированием в центре неё иона металла (чаще всего калия). Эти пластинки располагаются стопкой друг над другом[38].
На концах хромосом могут образовываться и другие структуры: основания могут быть расположены в одной цепочке или в разных параллельных цепочках. Кроме этих «стопочных» структур теломеры формируют большие петлеобразные структуры, называемые Т-петли или теломерные петли. В них одноцепочечная ДНК располагается в виде широкого кольца, стабилизированного теломерными белками[39]. В конце Т-петли одноцепочечная теломерная ДНК присоединяется к двухцепочечной ДНК, нарушая спаривание цепочек в этой молекуле и образуя связи с одной из цепей. Это трёхцепочечное образование называется Д-петля (от англ. displacement loop)[38].
Биологические функции
ДНК является носителем генетической информации, записанной в виде последовательности нуклеотидов с помощью генетического кода. С молекулами ДНК связаны два основополагающих свойства живых организмов — наследственность и изменчивость. В ходе процесса, называемого репликацией ДНК, образуются две копии исходной цепочки, наследуемые дочерними клетками при делении, таким образом образовавшиеся клетки оказываются генетически идентичны исходной.
Генетическая информация реализуется при экспрессии генов в процессах транскрипции (синтеза молекул РНК на матрице ДНК) и трансляции (синтеза белков на матрице РНК).
Последовательность нуклеотидов «кодирует» информацию о различных типах РНК: информационных, или матричных (иРНК), рибосомальных (рРНК) и транспортных (тРНК). Все эти типы РНК синтезируются на основе ДНК в процессе транскрипции. Роль их в биосинтезе белков (процессе трансляции) различна. Информационная РНК содержит информацию о последовательности аминокислот в белке, рибосомальные РНК служат основой для рибосом (сложных нуклеопротеиновых комплексов, основная функция которых — сборка белка из отдельных аминокислот на основе иРНК), транспортные РНК доставляют аминокислоты к месту сборки белков — в активный центр рибосомы, «ползущей» по иРНК.
Структура генома
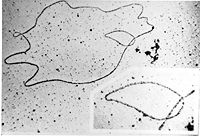
ДНК генома бактериофага: фотография под трансмиссионным электронным микроскопом.
Большинство природных ДНК имеет двухцепочечную структуру, линейную (эукариоты, некоторые вирусы и отдельные роды бактерий) или кольцевую (прокариоты, хлоропласты и митохондрии). Линейную одноцепочечную ДНК содержат некоторые вирусы и бактериофаги. В клетках эукариот ДНК располагается главным образом в ядре в виде набора хромосом. Бактериальная (прокариоты) ДНК обычно представлена одной кольцевой молекулой ДНК, расположенной в неправильной формы образовании в цитоплазме, называемым нуклеоидом [40]. Генетическая информация генома состоит из генов. Ген — единица передачи наследственной информации и участок ДНК, который влияет на определённую характеристику организма. Ген содержит открытую рамку считывания, которая транскрибируется, а также регуляторные последовательности, например, промотор и энхансер, которые контролируют экспрессию открытых рамок считывания.
У многих видов только малая часть общей последовательности генома кодирует белки. Так только около 1,5 % генома человека состоит из кодирующих белок экзонов, а больше 50 % ДНК человека состоит из некодирующих повторяющихся последовательностей ДНК[41]. Причины наличия такого большого количества некодирующей ДНК в эукариотических геномах и огромная разница в размерах геномов (С-значение) одна из неразрешённых научных загадок[42].
Последовательности генома, не кодирующие белок
-
Основная статья: Некодирующая ДНК
В настоящее время накапливается всё больше данных, противоречащих идее о некодирующих последовательностях как «мусорной ДНК» (англ. junk DNA). Теломеры и центромеры содержат малое число генов, но они важны для функционирования и стабильности хромосом[36][43]. Часто встречающаяся форма некодирующих последовательностей человека — псевдогены, копии генов, инактивированные в результате мутаций[44]. Эти последовательности нечто вроде молекулярных ископаемых, хотя иногда они могут служить исходным материалом для дупликации и последующей дивергенции генов[45]. Другой источник разнообразия белков в организме, это использование интронов в качестве «линий разреза и склеивания» в альтернативном сплайсинге[46]. Наконец, некодирующие белок последовательности могут кодировать вспомогательные клеточные РНК, например, мяРНК[47]. Недавнее исследование транскрипции генома человека показало, что 10 % генома даёт начало полиаденилированным РНК [48], а исследование и генома мыши показало, что 62 % его транскрибируется[49].
Транскрипция и трансляция
Генетическая информация, закодированная в ДНК, должна быть прочитана и в конечном итоге выражена в синтезе различных биополимеров, из которых состоят клетки. Последовательность оснований в цепочке ДНК напрямую определяет последовательность оснований в РНК, на которую она «переписывается» в процессе, называемом транскрипцией. В случае мРНК эта последовательность определяет аминокислоты белка. Соотношение между нуклеотидной последовательностью мРНК и аминокислотной последовательностью определяется правилами трансляции, которые называются генетическим кодом. Генетический код состоит из трёхбуквенных «слов», называемых кодонами, состоящих из трёх нуклеотидов (то есть ACT CAG TTT и т. п.). Во время транскрипции нуклеотиды гена копируются на синтезируемую РНК РНК-полимеразой. Эта копия в случае мРНК декодируется рибосомой, которая «читает» последовательность мРНК, осуществляя спаривание матричной РНК с транспортными РНК, которые присоединены к аминокислотам. Поскольку в трёхбуквенных комбинациях используются 4 основания, всего возможны 64 кодона (4³ комбинации). Кодоны кодируют 20 стандартных аминокислот, каждой из которых соответствует в большинстве случаев более одного кодона. Один из трёх кодонов, которые располагаются в конце мРНК, не означает аминокислоту и определяет конец белка, это «стоп» или «нонсенс» кодоны — TAA, TGA, TAG.
Репликация
Деление клеток необходимо для размножения одноклеточного и роста многоклеточного организма, но до деления клетка должна удвоить геном, чтобы дочерние клетки содержали ту же генетическую информацию, что и исходная клетка. Из нескольких теоретически возможных механизмов удвоения (репликации) ДНК реализуется полуконсервативный. Две цепочки разделяются и затем каждая недостающая комплементарная последовательность ДНК воспроизводится ферментом ДНК-полимеразой. Этот фермент строит полинуклеотидную цепь, находя правильное основание через комплементарное спаривание оснований и присоединяя его к растущей цепочке. ДНК-полимераза не может начинать новую цепь, а только лишь наращивать уже существующую, поэтому она нуждается в короткой цепочке нуклеотидов (праймере), синтезируемом праймазой. Так как ДНК-полимеразы могут строить цепочку только в направлении 5′ —> 3′, для копирования антипараллельных цепей используются разные механизмы[50].
Взаимодействие с белками
Взаимодействие ДНК с гистонами. Основные аминокислоты гистонов (на рисунке показаны голубым цветом) взаимодействуют с кислотными фосфатными группами ДНК (красный цвет).
Все функции ДНК зависят от её взаимодействия с белками. Взаимодействия могут быть как неспецифическими, когда белок присоединяется к любой молекуле ДНК или зависеть от наличия особой последовательности. Ферменты также могут взаимодействовать с ДНК, из них наиболее важные это РНК-полимеразы, которые копируют последовательность оснований ДНК на РНК в транскрипции или при синтезе новой цепи ДНК — репликации.
Структурные и регуляторные белки
Хорошо изученными примерами взаимодействия белков и ДНК, не зависящего от нуклеотидной последовательности ДНК, является взаимодействие со структурными белками. В клетке ДНК связана с этими белками, образуя компактную структуру, которая называется хроматин. У прокариот хроматин образован при присоединении к ДНК небольших щелочных белков — гистонов, менее упорядоченный хроматин прокариот содержит гистон-подобные белки[51][52]. Гистоны формируют дискообразную белковую структуру — нуклеосому, вокруг каждой из которых вмещается два оборота спирали ДНК. Неспецифические связи между гистонами и ДНК образуются за счёт ионных связей щелочных аминокислот гистонов и кислотных остатков сахарофостфатного остова ДНК[53]. Химические модификации этих аминокислот включают метилирование, фосфорилирование и ацетилирование[54]. Эти химические модификации изменяют силу взаимодействия между ДНК и гистонами, влияя на доступность специфических последовательностей для факторов транскрипции и изменяя скорость транскрипции[55]. Другие белки в составе хроматина, которые присоединяются к неспецифическим последовательностям — белки с высокой подвижностью в гелях, которые ассоциируют большей частью с согнутой ДНК[56]. Эти белки важны для образования в хроматине структур более высокого порядка[57]. Особая группа белков, присоединяющихся к ДНК это белки, которые ассоциируют с одноцепочечной ДНК. Наиболее хорошо охарактеризованный белок этой группы у человека — репликационный белок А, без которого невозможно протекание большинства процессов, где расплетается двойная спираль, включая репликацию, рекомбинацию, и репарацию. Белки этой группы стабилизируют одноцепочечную ДНК и предотвращают формирование стеблей-петель или деградации нуклеазами[58].
В то же время другие белки узнают и присоединяются к специфическим последовательностям. Наиболее изученная группа таких белков — различные классы факторов транскрипции, то есть белки, регулирующие транскрипцию. Каждый из этих белков узнаёт свою последовательность, часто в промоторе и активирует или подавляет транскрипцию гена. Это происходит при ассоциации факторов транскрипции с РНК-полимеразой либо напрямую, либо через белки-посредники. Полимераза ассоциирует сначала с белками, а потом начинает транскрипцию[59]. В других случаях факторы транскрипции могут присоединяться к ферментам, которые модифицируют находящиеся на промоторах гистоны, что изменяет доступность ДНК для полимераз[60].
Так как специфические последовательности встречаются во многих местах генома, изменения в активности одного типа фактора транскрипции может изменить активность тысяч генов[61]. Соответственно, эти белки часто регулируются в процессах ответа на изменения в окружающей среде, развития организма и дифференцировки клеток. Специфичность взаимодействия факторов транскрипции с ДНК обеспечивается многочисленными контактами между аминокислотами и основаниями ДНК, что позволяет им «читать» последовательность ДНК. Большинство контактов с основаниями происходит в главной бороздке, где основания более доступны[14].
Ферменты, модифицирующие ДНК
Топоизомеразы и хеликазы
В клетке ДНК находится в компактном т. н. суперскрученном состоянии, иначе она не смогла бы в ней уместится. Для протекания жизненноважных процессов ДНК должна быть раскручена, что производится двумя группами белков — топоизомеразами и хеликазами.
Топоизомеразы — ферменты, которые имеют и нуклеазную и лигазную активности. Эти белки изменяют степень суперскрученности в ДНК. Некоторые из этих ферментов разрезают спираль ДНК и позволяют вращаться одной из цепей, тем самым уменьшая уровень суперскрученности, после чего фермент заделывает разрыв[32]. Другие ферменты могут разрезать одну из цепей и проводить вторую цепь через разрыв, а потом лигировать разрыв в первой цепи[62]. Топоизомеразы необходимы во многих процессах, связанных с ДНК, таких как репликация и транкрипция[33].
Хеликазы — белки, которые являются одним из молекулярных моторов. Они используют химическую энергию нуклеотидтрифосфатов, чаще всего АТФ, для разрыва водородных связей между основаниями, раскручивая двойную спираль на отдельные цепочки[63]. Эти ферменты важны для большинства процессов, где белкам необходим доступ к основаниям ДНК.
Нуклеазы и лигазы
В различных процессах, происходящих в клетке, например, рекомбинации и репарации участвуют ферменты, способные разрезать и восстанавливать целостность нитей ДНК. Ферменты, разрезающие ДНК, носят название нуклеаз. Нуклеазы, которые гидролизуют нуклеотиды на концах молекулы ДНК называются экзонуклеазами, а эндонуклеазы разрезают ДНК внутри цепи. Наиболее часто используемые в молекулярной биологии и генетической инженерии нуклеазы — это рестриктазы, которые разрезают ДНК около специфических последовательностей. Например, фермент EcoRV (рестрикционный фермент № 5 из E. coli) узнаёт шестинуклеотидную последовательность 5′-GAT|ATC-3′ и разрезает ДНК в месте, указанном вертикальной линией. В природе эти ферменты защищают бактерии от заражения бактериофагами, разрезая ДНК фага, когда она вводится в бактериальную клетку. В этом случае нуклеазы — часть системы модификации-рестрикции[64]. ДНК-лигазы сшивают сахарофосфатные основания в молекуле ДНК, используя энергию АТФ. Рестрикционные нуклеазы и лигазы используются в клонировании и фингерпринтинге.
ДНК-полимераза I (кольцеобразная структура, состоящая из нескольких одинаковых молекул белка, показанных разными цветами), лигирующая повреждённую цепь ДНК
Полимеразы
Существует также важная для метаболизма ДНК группа ферментов, которые синтезируют цепи полинуклеотидов из нуклеозидтрифосфатов — ДНК-полимеразы. Они добавляют нуклеотиды к 3′-гидроксильной группе предыдущего нуклеотида в цепи ДНК, поэтому все полимеразы работают в направлении 5′—> 3′ [65]. В активном центре этих ферментов субстрат — нуклеозидтрифосфат — спаривается с комплементарным основанием в составе одноцепочечной полинулеотидной цепочки — матрицы.
В процессе репликации ДНК, ДНК-зависимая ДНК-полимераза синтезирует копию исходной последовательности ДНК. Точность очень важна в этом процессе, так как ошибки в полимеризации приведут к мутациям, поэтому многие полимеразы обладают способностью к «редактированию» — исправлению ошибок. Полимераза узнаёт ошибки в синтезе по отсутствию спаривания между неправильными нуклеотидами. После определения отсутствия спаривания активируется 3′—> 5′ экзонуклеазная активность полимеразы и неправильное основание удаляется[66]. В большинстве организмов ДНК-полимеразы работают в виде большого комплекса, называемого реплисомой, которая содержит многочисленные дополнительные субъединицы, например, хеликазы[67].
РНК-зависимые ДНК-полимеразы — специализированный тип полимераз, которые копируют последовательность РНК на ДНК. К этому типу относится вирусный фермент обратная транскриптаза, который используется ретровирусами при инфекции клеток, а также теломераза, необходимая для репликации теломер[68]. Теломераза — необычный фермент, потому что она содержит собственную матричную РНК[36].
Транскрипция осуществляется ДНК-зависимой РНК-полимеразой, которая копирует последовательность ДНК одной цепочки на мРНК. В начале транскрипции гена РНК-полимераза присоединяется к последовательности в начале гена, называемой промотором, и расплетает спираль ДНК. Потом она копирует последовательность гена на матричную РНК до тех пор, пока не дойдёт до участка ДНК в конце гена — терминатора, где она останавливается и отсоединяется от ДНК. Также как ДНК-зависимая ДНК-полимераза человека, РНК-полимераза II, которая транскрибирует большую часть генов в геноме человека, работает в составе большого белкового комплекса, содержащего регуляторные и дополнительные единицы [69].
Генетическая рекомбинация
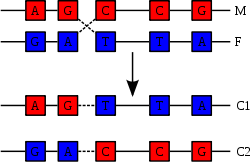
Рекомбинация происходит в результате физического разрыва в хромосомах (М) и (F) и их последующего соединения с образованием двух новых хромосом (C1 and C2)
Двойная спираль ДНК обычно не взаимодействует с другими сегментами ДНК, и в человеческих клетках разные хромосомы пространственно разделены в ядре[70]. Это расстояние между разными хромосомами важно для способности ДНК действовать в качестве стабильного носителя информации. В процессе рекомбинации с помощью ферментов две спирали ДНК разрываются, обмениваются участками, после чего непрерывность спиралей восстанавливается, поэтому обмен участками негомологичных хромосом может привести к повреждению целостности генетического материала.
Рекомбинация позволяет хромосомам обмениваться генетической информацией, в результате этого образуются новые комбинации генов, что увеличивает эффективность естественного отбора и важно для быстрой эволюции новых белков[71]. Генетическая рекомбинация также играет роль в репарации, особенно в ответе клетки на разрыв обеих цепей ДНК.[72]
Самая распространённая форма кроссинговера — это гомологичная рекомбинация, когда принимающие участие в рекомбинации хромосомы имеют очень похожие последовательности. Иногда в качестве участков гомологии выступают транспозоны. Негомологичная рекомбинация может привести к повреждению клетки, поскольку в результате такой рекомбинации возникают транслокации. Реакция рекомбинации катализируется ферментами, которые называются рекомбиназы, напрмер, Cre. На первом этапе реакции рекомбиназа делает разрыв в одной из цепей ДНК, позволяя этой цепи отделиться от комплементарной цепи и присоединится к одной из цепей второй хроматиды. Второй разрыв в цепи второй хроматиды позволяет ей также отделиться и присоединится к оставшейся без пары цепи из первой хроматиды, фомируя структуру Холлидея. Структура Холлидея может передвигаться вдоль соединённой пары хромосом, меняя цепи местами. Реакция рекомбинации завершается, когда фермент разрезает соединение, а две цепи лигируются.[73]
Эволюция метаболизма, основанного на ДНК
ДНК содержит генетическую информацию, которая делает возможной жизнедеятельность, рост, развитие и размножение всех современных организмов. Однако как долго в течение четырёх миллиардов лет истории жизни на Земле ДНК была главным носителем генетической информации, неизвестно. Существуют гипотезы, что РНК играла центральную роль в обмене веществ, поскольку она может и переносить генетическую информацию, и осуществлять катализ с помощью рибозимов[65][74] [75]. Кроме того, РНК — один из основных компонентов «фабрик белка» — рибосомы. Древний РНК-мир, где нуклеиновая кислота была использована и для катализа и для переноса информации мог послужить источником современного генетического кода, состоящего из четырёх оснований. Это могло произойти в результате того, что число оснований в организме было компромиссом между небольшим числом оснований, увеличивавшим точность репликации, и большим числом оснований, увеличивающим каталитическую активность рибозимов [76].
К сожалению, древние генетические системы не дошли до наших дней. ДНК в окружающей среде в среднем сохраняется в течение 1 миллиона лет, а потом деградирует до коротких фрагментов. Извлечение ДНК и определение последовательности их 16S рРНК генов из заключённых в кристаллах соли, образовавшихся 250 млн лет назад, бактериальных спор[77] служит темой оживлённой дискуссии в научной среде[78][79].
См. также
- Использование ДНК в технологии
- Вектор (биология)
- Генетическая генеалогия
- Действие излучений на структуру и функции ДНК
- Метилирование ДНК
- Мобильные элементы генома
- Мутация
- Нуклеопротеиды
- РНК
- Центральная догма молекулярной биологии
Ссылки
- Методы выделения и исследования ДНК
- Веб-адреса молекулярно-биологических журналов
- Международная база данных — последовательности ДНК из разных организмов (англ.).
- Репликация ДНК (анимация)(англ.)
- Веб-сайт Сэнгеровского Института одного из мировых лидеров в области определения последовательностей ДНК и их анализа (англ.)
- Software for forensic DNA Typing — eQMS::DNA
Рекомендуемая литература
- Альбертс Б.; Брей Д.; Льюис Дж. и др. Молекулярная биология клетки в 3-х томах. М.: Мир, 1994. 1558 с ISBN 5-03-001986-3
- Докинз Р. Эгоистичный ген, М.: Мир[80]
- История биологии с начала XX века до наших дней. М.: Наука, 1975. 660 с.
- Льюин Б. Гены. М.: Мир, 1987. 1064 с.
- Пташне М. Переключение генов. Регуляция генной активности и фаг лямбда. М.: Мир, 1989. 160 с,[81]
- Уотсон Дж. Д. Двойная спираль: воспоминания об открытии структуры ДНК. М.: Мир, 1969. 152 с.
Литература
- ↑ Dahm R (2005). «Friedrich Miescher and the discovery of DNA». Dev Biol 278 (2): 274–88. PMID 15680349.
- ↑ Hershey A, Chase M (1952). «Independent functions of viral protein and nucleic acid in growth of bacteriophage». J Gen Physiol 36 (1): 39–56. PMID 12981234.
- ↑ 1 2 Watson J, Crick F (1953). «Molecular structure of nucleic acids; a structure for deoxyribose nucleic acid». Nature 171 (4356): 737 – 8. PMID 13054692.
- ↑ The Nobel Prize in Physiology or Medicine 1962 Nobelprize .org Accessed 22 Dec 06
- ↑ Bruce Alberts Molecular Biology of the Cell; Fourth Edition. — New York and London: Garland Science. — ISBN ISBN 0-8153-3218-1
- ↑ Butler, John M. (2001) Forensic DNA Typing «Elsevier». pp. 14 — 15. ISBN 978-0-12-147951-0
- ↑ 1 2 Berg J., Tymoczko J. and Stryer L. (2002) Biochemistry. W. H. Freeman and Company ISBN 0-7167-4955-6
- ↑ Abbreviations and Symbols for Nucleic Acids, Polynucleotides and their Constituents IUPAC-IUB Commission on Biochemical Nomenclature (CBN) Accessed 03 Jan 2006
- ↑ Takahashi I, Marmur J. (1963). «Replacement of thymidylic acid by deoxyuridylic acid in the deoxyribonucleic acid of a transducing phage for Bacillus subtilis». Nature 197: 794 – 5. PMID 13980287.
- ↑ Agris P (2004). «Decoding the genome: a modified view». Nucleic Acids Res 32 (1): 223 – 38. PMID 14715921.
- ↑ Ghosh A, Bansal M (2003). «A glossary of DNA structures from A to Z». Acta Crystallogr D Biol Crystallogr 59 (Pt 4): 620 – 6. PMID 12657780.
- ↑ Mandelkern M, Elias J, Eden D, Crothers D (1981). «The dimensions of DNA in solution». J Mol Biol 152 (1): 153 – 61. PMID 7338906.
- ↑ Wing R, Drew H, Takano T, Broka C, Tanaka S, Itakura K, Dickerson R (1980). «Crystal structure analysis of a complete turn of B-DNA». Nature 287 (5784): 755 – 8. PMID 7432492.
- ↑ 1 2 Pabo C, Sauer R. «Protein-DNA recognition». Annu Rev Biochem 53: 293 – 321. PMID 6236744.
- ↑ Ponnuswamy P, Gromiha M (1994). «On the conformational stability of oligonucleotide duplexes and tRNA molecules». J Theor Biol 169 (4): 419–32. PMID 7526075.
- ↑ Clausen-Schaumann H, Rief M, Tolksdorf C, Gaub H (2000). «Mechanical stability of single DNA molecules». Biophys J 78 (4): 1997–2007. PMID 10733978.
- ↑ Chalikian T, Völker J, Plum G, Breslauer K (1999). «A more unified picture for the thermodynamics of nucleic acid duplex melting: a characterization by calorimetric and volumetric techniques». Proc Natl Acad Sci U S A 96 (14): 7853–8. PMID 10393911.
- ↑ Created from PDB 1JDG
- ↑ Klose R, Bird A (2006). «Genomic DNA methylation: the mark and its mediators». Trends Biochem Sci 31 (2): 89 – 97. PMID 16403636.
- ↑ Bird A (2002). «DNA methylation patterns and epigenetic memory». Genes Dev 16 (1): 6 – 21. PMID 11782440.
- ↑ Walsh C, Xu G. «Cytosine methylation and DNA repair». Curr Top Microbiol Immunol 301: 283 – 315. PMID 16570853.
- ↑ Gommers-Ampt J, Van Leeuwen F, de Beer A, Vliegenthart J, Dizdaroglu M, Kowalak J, Crain P, Borst P (1993). «beta-D-glucosyl-hydroxymethyluracil: a novel modified base present in the DNA of the parasitic protozoan T. brucei». Cell 75 (6): 1129 – 36. PMID 8261512.
- ↑ Douki T, Reynaud-Angelin A, Cadet J, Sage E (2003). «Bipyrimidine photoproducts rather than oxidative lesions are the main type of DNA damage involved in the genotoxic effect of solar UVA radiation». Biochemistry 42 (30): 9221 – 6. PMID 12885257.
- ↑ Cadet J, Delatour T, Douki T, Gasparutto D, Pouget J, Ravanat J, Sauvaigo S (1999). «Hydroxyl radicals and DNA base damage». Mutat Res 424 (1 – 2): 9 – 21. PMID 10064846.
- ↑ Shigenaga M, Gimeno C, Ames B (1989). «Urinary 8-hydroxy-2′-deoxyguanosine as a biological marker of in vivo oxidative DNA damage». Proc Natl Acad Sci U S A 86 (24): 9697 – 701. PMID 2602371.
- ↑ Cathcart R, Schwiers E, Saul R, Ames B (1984). «Thymine glycol and thymidine glycol in human and rat urine: a possible assay for oxidative DNA damage». Proc Natl Acad Sci U S A 81 (18): 5633 – 7. PMID 6592579.
- ↑ Ferguson L, Denny W (1991). «The genetic toxicology of acridines». Mutat Res 258 (2): 123 – 60. PMID 1881402.
- ↑ Jeffrey A (1985). «DNA modification by chemical carcinogens». Pharmacol Ther 28 (2): 237 – 72. PMID 3936066.
- ↑ Stephens T, Bunde C, Fillmore B (2000). «Mechanism of action in thalidomide teratogenesis». Biochem Pharmacol 59 (12): 1489 – 99. PMID 10799645.
- ↑ Braña M, Cacho M, Gradillas A, de Pascual-Teresa B, Ramos A (2001). «Intercalators as anticancer drugs». Curr Pharm Des 7 (17): 1745 – 80. PMID 11562309.
- ↑ Benham C, Mielke S (2005). «DNA mechanics». Annu Rev Biomed Eng 7: 21–53. PMID 16004565.
- ↑ 1 2 Champoux J (2001). «DNA topoisomerases: structure, function, and mechanism». Annu Rev Biochem 70: 369–413. PMID 11395412.
- ↑ 1 2 Wang J (2002). «Cellular roles of DNA topoisomerases: a molecular perspective». Nat Rev Mol Cell Biol 3 (6): 430–40. PMID 12042765.
- ↑ Created from NDB UD0017
- ↑ Greider C, Blackburn E (1985). «Identification of a specific telomere terminal transferase activity in Tetrahymena extracts». Cell 43 (2 Pt 1): 405–13. PMID 3907856.
- ↑ 1 2 3 Nugent C, Lundblad V (1998). «The telomerase reverse transcriptase: components and regulation». Genes Dev 12 (8): 1073–85. PMID 9553037.
- ↑ Wright W, Tesmer V, Huffman K, Levene S, Shay J (1997). «Normal human chromosomes have long G-rich telomeric overhangs at one end». Genes Dev 11 (21): 2801–9. PMID 9353250.
- ↑ 1 2 Burge S, Parkinson G, Hazel P, Todd A, Neidle S (2006). «Quadruplex DNA: sequence, topology and structure». Nucleic Acids Res 34 (19): 5402–15. PMID 17012276.
- ↑ Griffith J, Comeau L, Rosenfield S, Stansel R, Bianchi A, Moss H, de Lange T (1999). «Mammalian telomeres end in a large duplex loop». Cell 97 (4): 503–14. PMID 10338214.
- ↑ Thanbichler M, Wang S, Shapiro L (2005). «The bacterial nucleoid: a highly organized and dynamic structure». J Cell Biochem 96 (3): 506 – 21. PMID 15988757.
- ↑ Wolfsberg T, McEntyre J, Schuler G (2001). «Guide to the draft human genome». Nature 409 (6822): 824 – 6. PMID 11236998.
- ↑ Gregory T (2005). «The C-value enigma in plants and animals: a review of parallels and an appeal for partnership». Ann Bot (Lond) 95 (1): 133 – 46. PMID 15596463.
- ↑ Pidoux A, Allshire R (2005). «The role of heterochromatin in centromere function». Philos Trans R Soc Lond B Biol Sci 360 (1455): 569 – 79. PMID 15905142.
- ↑ Harrison P, Hegyi H, Balasubramanian S, Luscombe N, Bertone P, Echols N, Johnson T, Gerstein M (2002). «Molecular fossils in the human genome: identification and analysis of the pseudogenes in chromosomes 21 and 22». Genome Res 12 (2): 272 – 80. PMID 11827946.
- ↑ Harrison P, Gerstein M (2002). «Studying genomes through the aeons: protein families, pseudogenes and proteome evolution». J Mol Biol 318 (5): 1155 – 74. PMID 12083509.
- ↑ Soller M (2006). «Molecular fossils in the human genome: identification and analysis of the pseudogenes in chromosomes 21 and 22». Cell Mol Life Sci 63 (7-9): 796 – 819. PMID 16465448.
- ↑ Michalak P. (2006). «RNA world — the dark matter of evolutionary genomics» 19 (6): 1768 – 74. PMID 17040373.
- ↑ Cheng J, Kapranov P, Drenkow J, Dike S, Brubaker S et al. (2005). «RNA world — the dark matter of evolutionary genomics» 308: 1149 – 54. PMID 15790807.
- ↑ Mattick JS (2004). «RNA regulation: a new genetics?». Nat Rev Genet 5: 316-323. PMID 15131654.
- ↑ Albà M (2001). «Replicative DNA polymerases». Genome Biol 2 (1): REVIEWS3002. PMID 11178285.
- ↑ Sandman K, Pereira S, Reeve J (1998). «Diversity of prokaryotic chromosomal proteins and the origin of the nucleosome». Cell Mol Life Sci 54 (12): 1350 – 64. PMID 9893710.
- ↑ Dame RT (2005). «The role of nucleoid-associated proteins in the organization and compaction of bacterial chromatin». Mol. Microbiol. 56 (4): 858-70. PMID 15853876.
- ↑ Luger K, Mäder A, Richmond R, Sargent D, Richmond T (1997). «Crystal structure of the nucleosome core particle at 2.8 A resolution». Nature 389 (6648): 251 – 60. PMID 9305837.
- ↑ Jenuwein T, Allis C (2001). «Translating the histone code». Science 293 (5532): 1074 – 80. PMID 11498575.
- ↑ Ito T. «Nucleosome assembly and remodelling». Curr Top Microbiol Immunol 274: 1 – 22. PMID 12596902.
- ↑ Thomas J (2001). «HMG1 and 2: architectural DNA-binding proteins». Biochem Soc Trans 29 (Pt 4): 395 – 401. PMID 11497996.
- ↑ Grosschedl R, Giese K, Pagel J (1994). «HMG domain proteins: architectural elements in the assembly of nucleoprotein structures». Trends Genet 10 (3): 94–100. PMID 8178371.
- ↑ Iftode C, Daniely Y, Borowiec J (1999). «Replication protein A (RPA): the eukaryotic SSB». Crit Rev Biochem Mol Biol 34 (3): 141 – 80. PMID 10473346.
- ↑ Myers L, Kornberg R. «Mediator of transcriptional regulation». Annu Rev Biochem 69: 729 – 49. PMID 10966474.
- ↑ Spiegelman B, Heinrich R (2004). «Biological control through regulated transcriptional coactivators». Cell 119 (2): 157-67. PMID 15479634.
- ↑ Li Z, Van Calcar S, Qu C, Cavenee W, Zhang M, Ren B (2003). «A global transcriptional regulatory role for c-Myc in Burkitt’s lymphoma cells». Proc Natl Acad Sci U S A 100 (14): 8164 – 9. PMID 12808131.
- ↑ Schoeffler A, Berger J (2005). «Recent advances in understanding structure-function relationships in the type II topoisomerase mechanism». Biochem Soc Trans 33 (Pt 6): 1465 – 70. PMID 16246147.
- ↑ Tuteja N, Tuteja R (2004). «Unraveling DNA helicases. Motif, structure, mechanism and function». Eur J Biochem 271 (10): 1849–63. PMID 15128295.
- ↑ Bickle T, Krüger D (1993). «Biology of DNA restriction». Microbiol Rev 57 (2): 434 – 50. PMID 8336674.
- ↑ 1 2 Joyce C, Steitz T (1995). «Polymerase structures and function: variations on a theme?». J Bacteriol 177 (22): 6321 – 9. PMID 7592405.
- ↑ Hubscher U, Maga G, Spadari S. «Eukaryotic DNA polymerases». Annu Rev Biochem 71: 133 – 63. PMID 12045093.
- ↑ Johnson A, O’Donnell M. «Cellular DNA replicases: components and dynamics at the replication fork». Annu Rev Biochem 74: 283 – 315. PMID 15952889.
- ↑ Tarrago-Litvak L, Andréola M, Nevinsky G, Sarih-Cottin L, Litvak S (1994). «The reverse transcriptase of HIV-1: from enzymology to therapeutic intervention». FASEB J 8 (8): 497–503. PMID 7514143.
- ↑ Martinez E (2002). «Multi-protein complexes in eukaryotic gene transcription». Plant Mol Biol 50 (6): 925 – 47. PMID 12516863.
- ↑ Cremer T, Cremer C (2001). «Chromosome territories, nuclear architecture and gene regulation in mammalian cells». Nat Rev Genet 2 (4): 292-301. PMID 11283701.
- ↑ Pál C, Papp B, Lercher M (2006). «An integrated view of protein evolution». Nat Rev Genet 7 (5): 337 – 48. PMID 16619049.
- ↑ O’Driscoll M, Jeggo P (2006). «The role of double-strand break repair — insights from human genetics». Nat Rev Genet 7 (1): 45 – 54. PMID 16369571.
- ↑ Dickman M, Ingleston S, Sedelnikova S, Rafferty J, Lloyd R, Grasby J, Hornby D (2002). «The RuvABC resolvasome». Eur J Biochem 269 (22): 5492 – 501. PMID 12423347.
- ↑ Orgel L. «Prebiotic chemistry and the origin of the RNA world». Crit Rev Biochem Mol Biol 39 (2): 99 – 123. PMID 15217990.
- ↑ Davenport R (2001). «Ribozymes. Making copies in the RNA world». Science 292 (5520): 1278. PMID 11360970.
- ↑ Szathmáry E (1992). «What is the optimum size for the genetic alphabet?». Proc Natl Acad Sci U S A 89 (7): 2614 – 8. PMID 1372984.
- ↑ Vreeland R, Rosenzweig W, Powers D (2000). «Isolation of a 250 million-year-old halotolerant bacterium from a primary salt crystal». Nature 407 (6806): 897 – 900. PMID 11057666.
- ↑ Hebsgaard M, Phillips M, Willerslev E (2005). «Geologically ancient DNA: fact or artefact?». Trends Microbiol 13 (5): 212 – 20. PMID 15866038.
- ↑ Nickle D, Learn G, Rain M, Mullins J, Mittler J (2002). «Curiously modern DNA for a «250 million-year-old» bacterium». J Mol Evol 54 (1): 134 – 7. PMID 11734907.
- ↑ Эгоистичный ген в библиотеке FictionBook
- ↑ Все форумы > Книга «переключение генов» М. Пташне
|
Типы нуклеиновых кислот |
|
|---|---|
| Азотистые основания | Пурины (Аденин, Гуанин) | Пиримидины (Урацил, Тимин, Цитозин) |
| Нуклеозиды | Аденозин | Гуанозин | Уридин | Тимидин | Цитидин |
| Нуклеотиды | монофосфаты (АМФ, ГМФ, UMP, ЦМФ) | дифосфаты (АДФ, ГДФ, УДФ, ЦДФ) | трифосфаты (АТФ, ГТФ, УТФ, ЦТФ) | циклические (цАМФ, цГМФ, cADPR) |
| Рибонуклеиновые кислоты | РНК | мРНК | тРНК | рРНК | антисмысловые РНК | микроРНК | некодирующие РНК | piwi-interacting RNA | малые интерферирующие РНК | малые ядерные РНК | малые ядрышковые РНК | тмРНК |
| Дезоксирибонуклеиновые кислоты | ДНК | кДНК | Геном | msDNA | Митохондриальная ДНК |
| Аналоги нуклеиновых кислот | GNA | LNA | ПНК | TNA | Морфолино |
| Типы векторов | en:phagemid | Плазмиды | Фаг лямбда | en:cosmid | en:P1 phage | en:fosmid | BAC | YAC | HAC |
|
Основные группы биохимических молекул Аминокислоты · Пептиды · Белки · Углеводы · Нуклеотиды · Нуклеиновые кислоты · Липиды · Терпены · Каротиноиды · Стероиды · Флавоноиды · Алкалоиды · Гликозиды |
Wikimedia Foundation.
2010.
Генетические мутации — изменения в генах, которые вызывают неизлечимые заболевания. Но все ли мутации опасны? И почему такое вообще случается? Если у человека есть генетическая мутация, значит ли это, что у него проявится какая-то болезнь? Отвечаем на эти и другие вопросы в статье.
Содержание
- Что такое ДНК?
- Какова функция генов?
- Что такое мутация?
- Мутации: хорошие, плохие, нейтральные
- Что делать, если генетический тест показал мутацию?
Что такое ДНК?
Чтобы разобраться, что такое генетическая мутация, вспомним, как устроены ДНК и гены.
ДНК (дезоксирибонуклеиновая кислота) — это длинная молекула, которую принято называть «двойной спиралью». Она хранит биологическую информацию, которая «записана» в виде генетического кода.
Ген — это основная «единица» наследственной информации. Он представляет собой кусочек ДНК.
Какова функция генов?
В части генов в виде кода записаны «рецепты» изготовления белков. Именно белки выполняют основные функции для поддержания жизнедеятельности организма: они отвечают за пищеварение, кровообращение, иммунитет, передачу информации между клетками.
Код представляет собой последовательность нуклеотидов.
Нуклеотид — это конструкция, которая состоит из молекулы сахара, молекулы фосфата и основания.
В нашей ДНК есть четыре азотистых основания:
- аденин,
- гуанин,
- цитозин,
- тимин.
Основания одной цепи соединяются с основаниями другой цепи парами (аденин с тимином, цитозин с гуанином).
Если посмотреть на двойную спираль ДНК, то ее горизонтальные «ступени» будут парами оснований, а вертикальные боковые части — сахарами и фосфатами.
Чтобы изготовить белки по записанному в генах коду, специальные соединения — ферменты — «читают» и копируют код. В результате получаются длинные одноцепочечные молекулы — РНК (рибонуклеиновые кислоты), но это еще не белок. РНК лишь несут в себе информацию о первичной структуре белка, поэтому их называют матричными (сокращенно — мРНК). Эти молекулы покидают ядро клетки и перемещаются в ее цитоплазму. Там специальные органы — рибосомы — считывают код мРНК и изготавливают по этому «рецепту» белок.
Что такое мутация?
Генетическая мутация — это любое изменение в нуклеотидной последовательности ДНК.
К основным типам мутаций относятся:
- транзиция — замена аденина на гуанин или замена тимина на цитозин;
- трансверсия — аденин или гуанин меняются местами с тимином или цитозином;
- делеция — потеря участка ДНК;
- инсерция — добавление участка ДНК;
- дупликация — удвоение участка ДНК;
- инверсия — изменение, при котором участок хромосомы поворачивается на 180°;
- транслокация — мутация, при которой хромосомы обмениваются фрагментами.
Мутации могут происходить по разным причинам.
Спонтанные генетические мутации
Они происходят на протяжении всей нашей жизни. Можно сказать, что это нормальное явление, которое случается в ходе разных процессов, например, при копировании ДНК.
Как правило, такие ошибки не грозят серьезными последствиями, потому что у нашего организма есть механизмы защиты.
К ним относится, например, апоптоз — процесс программируемой гибели «испорченной» клетки, или репарация — починка нити ДНК. В этом случае ошибочный участок ДНК вырезается, а на его месте формируется новый.
Мутации, вызванные внешним влиянием
Мутации могут возникнуть под воздействием внешних неблагоприятных факторов, например, химических веществ, ионизирующего излучения или заражения вирусами.
Белки, которые отвечают за исправление ошибок, как правило, могут исправить испорченные цепи ДНК или привести одну хромосому в соответствие с другой. Но, если ошибки произошли на уровне генома или количества хромосом, защитные механизмы будут бессильны.
Наследственные генетические мутации
Такие мутации достаются человеку от родителей. Бывают случаи, когда генетическое нарушение передается из поколения в поколение (как, например, болезнь гемофилия), иногда мутации происходят в яйцеклетках и сперматозоидах и таким образом передаются ребенку.
Бывают случаи, когда мутации возникают на этапе формирования зиготы — клетки, которая образуется в результате оплодотворения. Как и в предыдущем случае, механизмы репарации с такими мутациями работают далеко не всегда, а ряд заболеваний и вовсе связан с нарушениями в процессе починки (например, пигментная ксеродерма — заболевание кожи, представляющее собой повышенную чувствительность к ультрафиолету).
Мутации: хорошие, плохие, нейтральные
Не все генетические мутации опасны. Важно понимать, что именно мутации объясняют генетические различия между видами. Изменения генов влекут за собой изменение характеристик организма, и в результате этого он может стать либо более, либо менее приспособленным к выживанию.
В ходе естественного отбора преимущество получают те живые существа, которые обладают более «полезным» набором характеристик, и тогда мутация закрепляется в популяции, становясь нормой.
«Хорошие» мутации
Ученым известно, что, например, у людей с определенным вариантом гена GPR75 риск ожирения снижен на 54%. А те, у кого есть хотя бы одна копия такого варианта гена, имеют более низкий индекс массы тела.
Мутации генов могут давать человеку и другие преимущества: так, мутировавший ген EPOR дал финскому лыжнику, трехкратному олимпийскому чемпиону Ээро Мянтюранта высокую чувствительность к эритропоэтину — гормону, который помогает нашим клеткам поддерживать оптимальный уровень кислорода и выводить углекислый газ. Это изменило и объем красных кровяных клеток в крови спортсмена, и объем кислорода, который эти клетки способны переносить. В результате Мянтюранта получил супервыносливость — его организм легко справлялся с повышенной потребностью в кислороде во время физических нагрузок.
«Плохие» мутации
Генетические мутации могут вызывать различные заболевания. Например, изменения гена DMD вызывают дистрофию Дюшенна — нервно-мышечное заболевание, которое проявляется у мужчин намного чаще, чем у женщин. А к серповидноклеточной анемии — нарушению в строении белка гемоглобина, который переносит кислород от легких к органам, — приводят мутации гена HBB. Хорея Гентингтона — тяжелое заболевание нервной системы — развивается из-за мутации в гене HTT.
Однако далеко не всегда генетическое заболевание связано с мутацией одного гена. Так, синдром Дауна возникает из-за изменения количества хромосом — в клетках пациентов с этой болезнью 47 хромосом вместо обычных 46.
Ряд заболеваний, таких как рак, диабет, расстройства аутического спектра, появляются из-за комбинации факторов. Пациенты могут иметь генетическую предрасположенность, но значительную роль играют и внешние факторы — неправильный образ жизни, неблагоприятная окружающая среда.
«Нейтральные» мутации
Нейтральные мутации, как следует из названия, не оказывают на здоровье человека ни положительного, ни отрицательного эффекта. Как правило, эти мутации затрагивают нуклеотидную последовательность ДНК, но не сказываются на строении белков.
Так происходит, потому что наш генетический код обладает так называемой избыточностью — это значит, что ряд аминокислот кодируется несколькими способами, чтобы случайные ошибки при копировании с меньшей вероятностью привели к нарушению функции или отсутствию кодируемого белка.
Бывает и так, что мутация гена все-таки меняет аминокислоту. Тем не менее, это не всегда приводит к нарушению функции белка.
Что делать, если генетический тест показал мутацию?
Во многих случаях наличие генетических нарушений не означает, что у человека непременно разовьется какое-то заболевание. Это значит лишь то, что пациент обладает генетическим вариантом, который чаще встречается у людей с этим заболеванием, и, вероятно, предрасположен к возникновению проблемы больше остальных.
Важнейшую роль в таких случаях играют внешние факторы: образ жизни, привычки, окружающая среда.
Ученые сходятся на том, что важными условиями сохранения здоровья являются:
● сбалансированное питание, богатое овощами и фруктами;
● регулярные занятия спортом;
● отказ от курения и алкоголя;
● достаточное количество сна.
Эти правила помогут значительно снизить риск развития таких распространенных заболеваний, как рак, проблемы с сердечно-сосудистой системой, диабет второго типа.
В том случае, если у человека есть мутация, связанная с моногенным заболеванием (то есть таким, которое возникает из-за «поломки» всего лишь одного гена), то существует риск, что он передаст этот вариант гена своему ребенку. Кроме того, болезнь может проявиться и у самого обладателя «плохого» гена — в этом случае ему следует обратиться к специалистам. Как правило, генетические заболевания не лечатся, но врач сможет порекомендовать препараты или изменения образа жизни (например, диета), чтобы уменьшить проявления болезни.
Результаты Генетического теста Атлас подскажут персональные рекомендации по улучшению образа жизни, которые помогут минимизировать риск появления заболеваний. Используя эти знания, будет проще спланировать подходящий рацион, спортивные нагрузки и тренировки, профилактические обследования.
Больше статей о генетике в блоге Атласа:
- Из чего состоит геном человека
- Кому и зачем нужны ДНК-банки
- Генная терапия: шанс или фантастика
- Very Well Health, What Are Genes, DNA, and Chromosomes? 2022
- Your Genome, What is a genome? 2017
- Medline Plus, What is DNA? 2021
- National Human Genome Research Institute, Mutation
Что такое ДНК, и из чего она состоит? Кто и когда открыл эту молекулу в клетках человека и других живых организмов? Чем уникален открытый учеными механизм наследования, и какие последствия ждал весь мир после этого открытия? Всю необходимую информацию Вы можете узнать, прочитав эту статью.

Когда впервые в истории появилось упоминание о ДНК
Иоганнес Фридрих Фишер – врач и биолог-исследователь родом из Швейцарии, стал первым в мире ученым, выделившим нуклеиновую кислоту. Открытие случилось в 1869 году, когда он занимался изучением животных клеток, а именно лейкоцитов, которых много содержалось в гное. Совершенно случайно молодой ученый заметил, что при отмывании лейкоцитов с гнойных повязок от них остается загадочное соединение. Под микроскопом Иоганн обнаружил, что оно содержится в ядрах клеток. Это соединение Мишер назвал нуклеином, а в процессе изучения его свойств переименовал в нуклеиновую кислоту, из-за наличия свойств, как у кислот.
Роль и функции только открытой нуклеиновой кислоты были неизвестны. Однако многие ученые того времени уже высказывали свои теории и предположения о существовании механизмов наследования.
Нынешние взгляды на состав молекулы ДНК ассоциируются у людей с именами английских ученых Джорджа Уотсона и Фрэнсиса Крика, которые открыли структуру данной молекулы в 1953 году. За несколько лет до этого, в тридцатые годы, ученые из советского союза А.Н. Белозерский и А.Р. Кезеля доказали наличие ДНК в клетках во всех живых организмах, тем самым они опровергли теорию о том, что молекула ДНК находится только в клетках животных, а в клетках растений присутствует только РНК. Лишь спустя несколько лет, в 1944 году, группой освальдских ученых было установлено, что молекула ДНК является механизмом сохранения наследственной информации клетки. Таким образом, благодаря совместным усилиям и трудам исследователей человечество познало тайну процесса эволюции и его основных принципов.
ДНК в медицине
Открытие состава молекулы дезоксирибонуклеиновой кислоты позволило перейти медицине на новый уровень развития. Появилось большое количество новых направлений практической медицины, стали доступны новые методы лечения, диагностики. Благодаря этому фундаментальному открытию для науки и современным технологиям, человечеству стали доступны:
- Возможность поставить диагноз на ранней стадии заболевания, когда оно еще находится в скрытом периоде, и никаких симптомов не проявляется.
- Тест ДНК на установление родственных связей у человека.
- Тесты на наличие у человека аллергии или непереносимости некоторых пищевых продуктов. Индивидуальные исследования помогут выявить, какая пища хорошо усваивается организмом, а какая плохо или вообще не усваивается, и что может стать причиной аллергической реакции у исследуемого.
- Анализ ДНК этнических особенностей. Возможность узнать, какие этносы формируют Вашу внешность, и из каких народов были Ваши далекие предки
- Тест на наличие врожденных заболеваний, передающиеся через поколения, оценка риска их возникновения у тестируемого человека.
И это еще не все доступные для людей услуги, которые может предложить медицина, изучающая генетику. Выше были представлены только самые популярные среди людей тесты. Перспективой для многих ученых-генетиков является создание таких лекарств, способных победить все болезни на Земле и даже смертность.
Строение молекулы ДНК
Молекула ДНК состоит из органических соединений — нуклеотидов, которые скручиваются в две спиралевидные цепи. Нуклеотиды в этих цепях – это базовые элементы, с помощью которых потом будут кодироваться и выстраиваться гены. В составе одного гена возможны несколько вариантов расположения некоторых нуклеотидов, поэтому вместе с тем, как меняется структура гена, меняется и его функциональность.
От цепочки к хромосоме
В каждом живом организме находится миллионы клеток, а внутри этих клеток находится ядро. Клетки, содержащие в себе ядро, называются эукариотами или ядерными. У древних одноклеточных нет оформленного ядра. К таким безъядерным одноклеточным, или прокариотам, относятся бактерии и археи, например, кишечная палочка или серая анаэробная бактерия. Также ядро отсутствует в клетках вирусов и вироидов, однако причисление вирусов к живым организмам – вопрос спорный, о котором по сей день дискуссируют ученые.
В ядре находятся хромосомы – структурный элемент, в котором содержится молекула ДНК в виде спирали, хранящая внутри себя всю генетическую информацию клетки.
Процесс упаковки ДНК спиралей
Количество нуклеотидов в ДНК велико, и нужны длинные цепочки, чтобы вместить все их число, поэтому нити ДНК закручиваются в две спирали, что позволяет укоротить цепочки в 5 раз, сделав их более компактными. Нити ДНК могут также закручиваться в форму суперспирали. Двойная спираль пересекает свою ось и накручивается на специальные гистоновые белки – гиразы, образуя при этом супервитки. Таким образом, двойная спираль закручивается в спираль более высокого порядка. Сокращение цепочек в этом случае произойдет в 30 раз.
Как гены связаны с ДНК
Ген – самый изученный на сегодняшний день участок ДНК. Гены являются структурной единицей наследственности всех живых организмов. Цепочки нуклеотидов в ДНК состоят из генов, которые определяют генотип особи, например, цвет и разрез глаз, тип кожи, рост, группу и резус фактор крови и другие физиологические качества и особенности внешности.
Еще много отраслей генетики до конца не изучены, и до конца не раскрыты все функции генома, но ученые до сих пор продолжают изучение генов, чтобы добиться новых открытий в области генетики.
Хромосома: определение и описание

Хромосомы – структурный элемент клетки, находящийся внутри ядра. Они содержат в себе молекулы ДНК, в которых содержится вся наследственная информация.
Строение и виды хромосом:
Хромосома состоит из двух «палочек» — хроматид, перетянутых по центру первичной перетяжкой – центромерой. Конец хромосомы называется теломером. Центромера может делить хромосому на короткое и длинное плечо.
Отсюда возникают различные типы хромосом:
- Равноплечая – центромера перетягивает хроматиды точно посередине;
- Неравноплечая – центромера неточно перетягивает хроматиды, из-за чего одно плечо хромосомы будет длиннее, а другое – короче. К этому типу относится Y-хромосома;
- Палочковидная – центромера перетягивает хроматиды практически на их концах, из-за чего по форме хромосома напоминает палочку;
- Точковые – очень мелкие хромосомы, форму которых трудно определить. В науке существуют 3 основные формы хромосом:
- Х-хромосома, встречающаяся у особей женского и мужского пола;
- Y-хромосома, встречающаяся только у мужских особей;
- В-хромосома, которая очень редко встречается в клетках растений. Обычно их число доходит до 6, редко – до 12. Ее наличие обуславливает различные болезни и побочные эффекты в организме
Всего в клетке человека находится 46 хромосом: 22 пары аутосом, встречающиеся у обоих полов, и одна пара половых хромосом: XY – у мужчин, XX – у женщин. Забавно, что если прибавить к количеству хромосом хотя бы одну пару, то человек мог бы быть шимпанзе или тараканом, а если отнять, то – кроликом.
Еще интересно то, что человек и ясень имеют одинаковое количество хромосом, несмотря на принадлежность к разным видам и царствам.
Наследственные болезни
Генетический код – система записи генетической информации в ДНК и РНК в виде определенной последовательности в цепочке нуклеотидов. Он должен сохранять наследственную информацию в первоначальном виде, восстанавливая повреждения цепочки в последующем поколении с помощью ДНК. Однако ген может каким-то образом быть поврежден, либо в нем может произойти мутация.
Генные мутации – изменение в последовательности нуклеотидов, например выпадение, замена, вставка другого нуклеотида в цепочку. Последствия этих мутаций могут быть полезные, вредные или нейтральные. Примером полезных мутаций является устойчивость к минусовым температурам, увеличенная плотность костей, меньшая потребность во сне, устойчивость к ВИЧ и другие. Примером вредных мутаций является аллергия на солнечный свет, глухота слепота и так далее. К нейтральным мутациям относятся те мутации, которые не влияют на жизнеспособность, например, гетерохромия.
Существуют также летальные и полулетальные мутации. Летальные мутации несовместимы с жизнью и приводят к гибели организма на ранних этапах его развития, например, при рождении у особи отсутствует головной мозг. Полулетальные мутации не приводят к смерти особи, но значительно уменьшают ее жизнеспособность. К таким мутациям относятся заболевания человека, передающиеся по наследству. Например, наличие 47-й хромосомы может вызвать у человека синдром Дауна, а, наоборот, отсутствие 46-й парной хромосомы – сидром Шерешевского-Тернера.
Расшифровка цепочки ДНК
Расшифровка цепочки ДНК в клетке – это исследование всех известных генов в клетках человека. Хоть цена за такую услугу значительно упала за последние десять лет, однако такое исследование по-прежнему остается дорогим удовольствием, и не каждый человек сможет позволить себе оплатить такую услугу. Чтобы уменьшить цену этого исследования, расшифровку ДНК стали делить по тематикам. Таким образом, появились различные тесты, которые исследуют интересующую человека группу генов и ее функции.
Как происходит расшифровка цепочки ДНК?
- Взятые на пробу образцы ДНК нагревают, чтобы двойная спираль раскрутилась и распалась на две нити.
- К интересующему участку цепочки генов прилепляется полимераза — фермент, синтезирующий полимеры нуклеиновых кислот. Процедура проходит при низких температурах.
- С помощью полимеразы в интересующих участков происходит синтезов генов, необходимых для изучения.
- Участки пропитывают светящейся краской, которая светится при лазерном воздействии.
Таким образом, ученые получают картину гена, которую можно изучить и расшифровать. Синтез РНК Нуклеотиды делятся на четыре базовых элемента, служащими основой для формирования генов: АТГЦ, или аденин, тимин, гуанин, цитозин. В их состав входят фосфорные остатки, азотистые основания и пептоза.
В ДНК эти нуклеотиды располагаются строго по парам параллельно друг другу строгими парами: аденин — с тимином, гуанин — с цитозином.
Важно, что молекула дезоксирибонуклеиновой кислоты не должна выходить за пределы мембраны ядра. С помощью РНК, которая играет роль копии участка цепи с генетическим кодом, генетическая цепочка может покинуть ядро, попасть вовнутрь клетки и воздействовать на ее внутренние процессы.
Как это происходит:
- Один конец генной спирали раскручивается, формируя две развернутые нити с цепочкой генов.
- К развернутому участку спирали подходит специальный фермент-строитель и поверх этого участка синтезирует его копию.
- У копии в структуре нуклеотидов тимин во всех парах заменяется на урацил, что позволяет копии генетической цепи покинуть ядро клетки. Синтез белка при помощи генов Основное взаимодействие, происходящее между генами и клеткой, состоит в том, что различные гены могут заставлять клетку производить синтез разных белков с самыми непредсказуемыми свойствами.
Итак, группа генов, участвующих в процессе старения клеток может, как заставить процесс старения идти быстрее, так и вовсе его остановить и запустить процесс омолаживания. То есть, каждый из генов может спровоцировать синтез нескольких видов белка.
Сутягина Дарья Сергеевна
Эксперт-генетик
В нашей ДНК содержится очень много информации, но пока мы можем расшифровать лишь небольшой процент генов. Добавлю несколько интересных фактов о ДНК: возможность двойной ДНК у человека. Такое явление случается, когда при беременности в утробе развиваются близнецы, но в процессе развития плода они сливаются в одного человека. Длина одной молекулы ДНК человека равна 2 метрам, а общая длина цепочки ДНК всех клеток тела человека равна 16 млрд. километрам, что равно расстоянию от Земли до Плутона. ДНК человека и кенгуру всего лишь 150 млн. лет назад были одинаковыми. Все знания и информация во всем мире могла бы уместиться всего лишь в 2 граммах дезоксирибонуклеиновой кислоты.

Two complementary regions of nucleic acid molecules will bind and form a double helical structure held together by base pairs.
In molecular biology, the term double helix[1] refers to the structure formed by double-stranded molecules of nucleic acids such as DNA. The double helical structure of a nucleic acid complex arises as a consequence of its secondary structure, and is a fundamental component in determining its tertiary structure. The term entered popular culture with the publication in 1968 of The Double Helix: A Personal Account of the Discovery of the Structure of DNA by James Watson.
The DNA double helix biopolymer of nucleic acid is held together by nucleotides which base pair together.[2] In B-DNA, the most common double helical structure found in nature, the double helix is right-handed with about 10–10.5 base pairs per turn.[3] The double helix structure of DNA contains a major groove and minor groove. In B-DNA the major groove is wider than the minor groove.[2] Given the difference in widths of the major groove and minor groove, many proteins which bind to B-DNA do so through the wider major groove.[4]
History[edit]
The double-helix model of DNA structure was first published in the journal Nature by James Watson and Francis Crick in 1953,[5] (X,Y,Z coordinates in 1954[6]) based on the work of Rosalind Franklin and her student Raymond Gosling, who took the crucial X-ray diffraction image of DNA labeled as «Photo 51», [7][8] and Maurice Wilkins, Alexander Stokes, and Herbert Wilson,[9] and base-pairing chemical and biochemical information by Erwin Chargaff.[10][11][12][13][14][15] The prior model was triple-stranded DNA.[16]
The realization that the structure of DNA is that of a double-helix elucidated the mechanism of base pairing by which genetic information is stored and copied in living organisms and is widely considered one of the most important scientific discoveries of the 20th century. Crick, Wilkins, and Watson each received one-third of the 1962 Nobel Prize in Physiology or Medicine for their contributions to the discovery.[17]
Nucleic acid hybridization[edit]
Hybridization is the process of complementary base pairs binding to form a double helix. Melting is the process by which the interactions between the strands of the double helix are broken, separating the two nucleic acid strands. These bonds are weak, easily separated by gentle heating, enzymes, or mechanical force. Melting occurs preferentially at certain points in the nucleic acid.[18] T and A rich regions are more easily melted than C and G rich regions. Some base steps (pairs) are also susceptible to DNA melting, such as T A and T G.[19] These mechanical features are reflected by the use of sequences such as TATA at the start of many genes to assist RNA polymerase in melting the DNA for transcription.
Strand separation by gentle heating, as used in polymerase chain reaction (PCR), is simple, providing the molecules have fewer than about 10,000 base pairs (10 kilobase pairs, or 10 kbp). The intertwining of the DNA strands makes long segments difficult to separate.[20] The cell avoids this problem by allowing its DNA-melting enzymes (helicases) to work concurrently with topoisomerases, which can chemically cleave the phosphate backbone of one of the strands so that it can swivel around the other.[21] Helicases unwind the strands to facilitate the advance of sequence-reading enzymes such as DNA polymerase.[22]
Base pair geometry[edit]

The geometry of a base, or base pair step can be characterized by 6 coordinates: shift, slide, rise, tilt, roll, and twist. These values precisely define the location and orientation in space of every base or base pair in a nucleic acid molecule relative to its predecessor along the axis of the helix. Together, they characterize the helical structure of the molecule. In regions of DNA or RNA where the normal structure is disrupted, the change in these values can be used to describe such disruption.
For each base pair, considered relative to its predecessor, there are the following base pair geometries to consider:[23][24][25]
- Shear
- Stretch
- Stagger
- Buckle
- Propeller: rotation of one base with respect to the other in the same base pair.
- Opening
- Shift: displacement along an axis in the base-pair plane perpendicular to the first, directed from the minor to the major groove.
- Slide: displacement along an axis in the plane of the base pair directed from one strand to the other.
- Rise: displacement along the helix axis.
- Tilt: rotation around the shift axis.
- Roll: rotation around the slide axis.
- Twist: rotation around the rise axis.
- x-displacement
- y-displacement
- inclination
- tip
- pitch: the height per complete turn of the helix.
Rise and twist determine the handedness and pitch of the helix. The other coordinates, by contrast, can be zero. Slide and shift are typically small in B-DNA, but are substantial in A- and Z-DNA. Roll and tilt make successive base pairs less parallel, and are typically small.
Note that «tilt» has often been used differently in the scientific literature, referring to the deviation of the first, inter-strand base-pair axis from perpendicularity to the helix axis. This corresponds to slide between a succession of base pairs, and in helix-based coordinates is properly termed «inclination».
Helix geometries[edit]
At least three DNA conformations are believed to be found in nature, A-DNA, B-DNA, and Z-DNA. The B form described by James Watson and Francis Crick is believed to predominate in cells.[26] It is 23.7 Å wide and extends 34 Å per 10 bp of sequence. The double helix makes one complete turn about its axis every 10.4–10.5 base pairs in solution. This frequency of twist (termed the helical pitch) depends largely on stacking forces that each base exerts on its neighbours in the chain. The absolute configuration of the bases determines the direction of the helical curve for a given conformation.
A-DNA and Z-DNA differ significantly in their geometry and dimensions to B-DNA, although still form helical structures. It was long thought that the A form only occurs in dehydrated samples of DNA in the laboratory, such as those used in crystallographic experiments, and in hybrid pairings of DNA and RNA strands, but DNA dehydration does occur in vivo, and A-DNA is now known to have biological functions. Segments of DNA that cells have methylated for regulatory purposes may adopt the Z geometry, in which the strands turn about the helical axis the opposite way to A-DNA and B-DNA. There is also evidence of protein-DNA complexes forming Z-DNA structures.
Other conformations are possible; A-DNA, B-DNA, C-DNA, E-DNA,[27] L-DNA (the enantiomeric form of D-DNA),[28] P-DNA,[29] S-DNA, Z-DNA, etc. have been described so far.[30] In fact, only the letters F, Q, U, V, and Y are now available to describe any new DNA structure that may appear in the future.[31][32] However, most of these forms have been created synthetically and have not been observed in naturally occurring biological systems.[citation needed] There are also triple-stranded DNA forms and quadruplex forms such as the G-quadruplex and the i-motif.

The structures of A-, B-, and Z-DNA.

The helix axis of A-, B-, and Z-DNA.
| Geometry attribute | A-DNA | B-DNA | Z-DNA |
|---|---|---|---|
| Helix sense | right-handed | right-handed | left-handed |
| Repeating unit | 1 bp | 1 bp | 2 bp |
| Rotation/bp | 32.7° | 34.3° | 60°/2 |
| bp/turn | 11 | 10.5 | 12 |
| Inclination of bp to axis | +19° | −1.2° | −9° |
| Rise/bp along axis | 2.3 Å (0.23 nm) | 3.32 Å (0.332 nm) | 3.8 Å (0.38 nm) |
| Pitch/turn of helix | 28.2 Å (2.82 nm) | 33.2 Å (3.32 nm) | 45.6 Å (4.56 nm) |
| Mean propeller twist | +18° | +16° | 0° |
| Glycosyl angle | anti | anti | C: anti, G: syn |
| Sugar pucker | C3′-endo | C2′-endo | C: C2′-endo, G: C2′-exo |
| Diameter | 23 Å (2.3 nm) | 20 Å (2.0 nm) | 18 Å (1.8 nm) |
Grooves[edit]

Major and minor grooves of DNA. Minor groove is a binding site for the dye Hoechst 33258.
Twin helical strands form the DNA backbone. Another double helix may be found by tracing the spaces, or grooves, between the strands. These voids are adjacent to the base pairs and may provide a binding site.[36] As the strands are not directly opposite each other, the grooves are unequally sized. One groove, the major groove, is 22 Å wide and the other, the minor groove, is 12 Å wide.[37] The narrowness of the minor groove means that the edges of the bases are more accessible in the major groove. As a result, proteins like transcription factors that can bind to specific sequences in double-stranded DNA usually make contacts to the sides of the bases exposed in the major groove.[4] This situation varies in unusual conformations of DNA within the cell (see below), but the major and minor grooves are always named to reflect the differences in size that would be seen if the DNA is twisted back into the ordinary B form.[38]
Non-double helical forms[edit]
Alternative non-helical models were briefly considered in the late 1970s as a potential solution to problems in DNA replication in plasmids and chromatin. However, the models were set aside in favor of the double-helical model due to subsequent experimental advances such as X-ray crystallography of DNA duplexes and later the nucleosome core particle, and the discovery of topoisomerases. Also, the non-double-helical models are not currently accepted by the mainstream scientific community.[39][40]
Bending[edit]
DNA is a relatively rigid polymer, typically modelled as a worm-like chain. It has three significant degrees of freedom; bending, twisting, and compression, each of which cause certain limits on what is possible with DNA within a cell. Twisting-torsional stiffness is important for the circularisation of DNA and the orientation of DNA bound proteins relative to each other and bending-axial stiffness is important for DNA wrapping and circularisation and protein interactions. Compression-extension is relatively unimportant in the absence of high tension.
Persistence length, axial stiffness[edit]
| Sequence | Persistence length / base pairs |
|---|---|
| Random | 154±10 |
| (CA)repeat | 133±10 |
| (CAG)repeat | 124±10 |
| (TATA)repeat | 137±10 |
DNA in solution does not take a rigid structure but is continually changing conformation due to thermal vibration and collisions with water molecules, which makes classical measures of rigidity impossible to apply. Hence, the bending stiffness of DNA is measured by the persistence length, defined as:
{{quote|The length of DNA over which the time-averaged orientation of the polymer becomes uncorrelated by a factor of e.[41]
This value may be directly measured using an atomic force microscope to directly image DNA molecules of various lengths. In an aqueous solution, the average persistence length is 46–50 nm or 140–150 base pairs (the diameter of DNA is 2 nm), although can vary significantly. This makes DNA a moderately stiff molecule.
The persistence length of a section of DNA is somewhat dependent on its sequence, and this can cause significant variation. The variation is largely due to base stacking energies and the residues which extend into the minor and major grooves.
Models for DNA bending[edit]
| Step | Stacking ΔG /kcal mol−1 |
|---|---|
| T A | -0.19 |
| T G or C A | -0.55 |
| C G | -0.91 |
| A G or C T | -1.06 |
| A A or T T | -1.11 |
| A T | -1.34 |
| G A or T C | -1.43 |
| C C or G G | -1.44 |
| A C or G T | -1.81 |
| G C | -2.17 |
At length-scales larger than the persistence length, the entropic flexibility of DNA is remarkably consistent with standard polymer physics models, such as the Kratky-Porod worm-like chain model.[43] Consistent with the worm-like chain model is the observation that bending DNA is also described by Hooke’s law at very small (sub-piconewton) forces. For DNA segments less than the persistence length, the bending force is approximately constant and behaviour deviates from the worm-like chain predictions.
This effect results in unusual ease in circularising small DNA molecules and a higher probability of finding highly bent sections of DNA.[44]
Bending preference[edit]
DNA molecules often have a preferred direction to bend, i.e., anisotropic bending. This is, again, due to the properties of the bases which make up the DNA sequence — a random sequence will have no preferred bend direction, i.e., isotropic bending.
Preferred DNA bend direction is determined by the stability of stacking each base on top of the next. If unstable base stacking steps are always found on one side of the DNA helix then the DNA will preferentially bend away from that direction. As bend angle increases then steric hindrances and ability to roll the residues relative to each other also play a role, especially in the minor groove. A and T residues will be preferentially be found in the minor grooves on the inside of bends. This effect is particularly seen in DNA-protein binding where tight DNA bending is induced, such as in nucleosome particles. See base step distortions above.
DNA molecules with exceptional bending preference can become intrinsically bent. This was first observed in trypanosomatid kinetoplast DNA. Typical sequences which cause this contain stretches of 4-6 T and A residues separated by G and C rich sections which keep the A and T residues in phase with the minor groove on one side of the molecule. For example:
| ¦ | ¦ | ¦ | ¦ | ¦ | ¦ | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| G | A | T | T | C | C | C | A | A | A | A | A | T | G | T | C | A | A | A | A | A | A | T | A | G | G | C | A | A | A | A | A | A | T | G | C | C | A | A | A | A | A | A | T | C | C | C | A | A | A | C |
The intrinsically bent structure is induced by the ‘propeller twist’ of base pairs relative to each other allowing unusual bifurcated Hydrogen-bonds between base steps. At higher temperatures this structure is denatured, and so the intrinsic bend is lost.
All DNA which bends anisotropically has, on average, a longer persistence length and greater axial stiffness. This increased rigidity is required to prevent random bending which would make the molecule act isotropically.
Circularization[edit]
DNA circularization depends on both the axial (bending) stiffness and torsional (rotational) stiffness of the molecule. For a DNA molecule to successfully circularize it must be long enough to easily bend into the full circle and must have the correct number of bases so the ends are in the correct rotation to allow bonding to occur. The optimum length for circularization of DNA is around 400 base pairs (136 nm)[citation needed], with an integral number of turns of the DNA helix, i.e., multiples of 10.4 base pairs. Having a non integral number of turns presents a significant energy barrier for circularization, for example a 10.4 x 30 = 312 base pair molecule will circularize hundreds of times faster than 10.4 x 30.5 ≈ 317 base pair molecule.[45]
The bending of short circularized DNA segments is non-uniform. Rather, for circularized DNA segments less than the persistence length, DNA bending is localised to 1-2 kinks that form preferentially in AT-rich segments. If a nick is present, bending will be localised to the nick site.[44]
Stretching[edit]
Elastic stretching regime[edit]
Longer stretches of DNA are entropically elastic under tension. When DNA is in solution, it undergoes continuous structural variations due to the energy available in the thermal bath of the solvent. This is due to the thermal vibration of the molecule combined with continual collisions with water molecules. For entropic reasons, more compact relaxed states are thermally accessible than stretched out states, and so DNA molecules are almost universally found in a tangled relaxed layouts. For this reason, one molecule of DNA will stretch under a force, straightening it out. Using optical tweezers, the entropic stretching behavior of DNA has been studied and analyzed from a polymer physics perspective, and it has been found that DNA behaves largely like the Kratky-Porod worm-like chain model under physiologically accessible energy scales.
Phase transitions under stretching[edit]
Under sufficient tension and positive torque, DNA is thought to undergo a phase transition with the bases splaying outwards and the phosphates moving to the middle. This proposed structure for overstretched DNA has been called P-form DNA, in honor of Linus Pauling who originally presented it as a possible structure of DNA.[29]
Evidence from mechanical stretching of DNA in the absence of imposed torque points to a transition or transitions leading to further structures which are generally referred to as S-form DNA. These structures have not yet been definitively characterised due to the difficulty of carrying out atomic-resolution imaging in solution while under applied force although many computer simulation studies have been made (for example,[46][47]).
Proposed S-DNA structures include those which preserve base-pair stacking and hydrogen bonding (GC-rich), while releasing extension by tilting, as well as structures in which partial melting of the base-stack takes place, while base-base association is nonetheless overall preserved (AT-rich).
Periodic fracture of the base-pair stack with a break occurring once per three bp (therefore one out of every three bp-bp steps) has been proposed as a regular structure which preserves planarity of the base-stacking and releases the appropriate amount of extension,[48] with the term «Σ-DNA» introduced as a mnemonic, with the three right-facing points of the Sigma character serving as a reminder of the three grouped base pairs. The Σ form has been shown to have a sequence preference for GNC motifs which are believed under the GNC hypothesis to be of evolutionary importance.[49]
Supercoiling and topology[edit]

Supercoiled structure of circular DNA molecules with low writhe. The helical aspect of the DNA duplex is omitted for clarity.
The B form of the DNA helix twists 360° per 10.4-10.5 bp in the absence of torsional strain. But many molecular biological processes can induce torsional strain. A DNA segment with excess or insufficient helical twisting is referred to, respectively, as positively or negatively supercoiled. DNA in vivo is typically negatively supercoiled, which facilitates the unwinding (melting) of the double-helix required for RNA transcription.
Within the cell most DNA is topologically restricted. DNA is typically found in closed loops (such as plasmids in prokaryotes) which are topologically closed, or as very long molecules whose diffusion coefficients produce effectively topologically closed domains. Linear sections of DNA are also commonly bound to proteins or physical structures (such as membranes) to form closed topological loops.
Francis Crick was one of the first to propose the importance of linking numbers when considering DNA supercoils. In a paper published in 1976, Crick outlined the problem as follows:
In considering supercoils formed by closed double-stranded molecules of DNA certain mathematical concepts, such as the linking number and the twist, are needed. The meaning of these for a closed ribbon is explained and also that of the writhing number of a closed curve. Some simple examples are given, some of which may be relevant to the structure of chromatin.[50]
Analysis of DNA topology uses three values:
- L = linking number — the number of times one DNA strand wraps around the other. It is an integer for a closed loop and constant for a closed topological domain.
- T = twist — total number of turns in the double stranded DNA helix. This will normally tend to approach the number of turns that a topologically open double stranded DNA helix makes free in solution: number of bases/10.5, assuming there are no intercalating agents (e.g., ethidium bromide) or other elements modifying the stiffness of the DNA.
- W = writhe — number of turns of the double stranded DNA helix around the superhelical axis
- L = T + W and ΔL = ΔT + ΔW
Any change of T in a closed topological domain must be balanced by a change in W, and vice versa. This results in higher order structure of DNA. A circular DNA molecule with a writhe of 0 will be circular. If the twist of this molecule is subsequently increased or decreased by supercoiling then the writhe will be appropriately altered, making the molecule undergo plectonemic or toroidal superhelical coiling.
When the ends of a piece of double stranded helical DNA are joined so that it forms a circle the strands are topologically knotted. This means the single strands cannot be separated any process that does not involve breaking a strand (such as heating). The task of un-knotting topologically linked strands of DNA falls to enzymes termed topoisomerases. These enzymes are dedicated to un-knotting circular DNA by cleaving one or both strands so that another double or single stranded segment can pass through. This un-knotting is required for the replication of circular DNA and various types of recombination in linear DNA which have similar topological constraints.
The linking number paradox[edit]
For many years, the origin of residual supercoiling in eukaryotic genomes remained unclear. This topological puzzle was referred to by some as the «linking number paradox».[51] However, when experimentally determined structures of the nucleosome displayed an over-twisted left-handed wrap of DNA around the histone octamer,[52][53] this paradox was considered to be solved by the scientific community.
See also[edit]
- Comparison of nucleic acid simulation software
- DNA nanotechnology
- G-quadruplex
- Molecular models of DNA
- Molecular structure of Nucleic Acids (publication)
- Non-B database
- Triple-stranded DNA
References[edit]
- ^ Kabai, Sándor (2007). «Double Helix». The Wolfram Demonstrations Project.
- ^ a b Alberts; et al. (1994). The Molecular Biology of the Cell. New York: Garland Science. ISBN 978-0-8153-4105-5.
- ^ Wang JC (1979). «Helical repeat of DNA in solution». PNAS. 76 (1): 200–203. Bibcode:1979PNAS…76..200W. doi:10.1073/pnas.76.1.200. PMC 382905. PMID 284332.
- ^ a b Pabo C, Sauer R (1984). «Protein-DNA recognition». Annu Rev Biochem. 53: 293–321. doi:10.1146/annurev.bi.53.070184.001453. PMID 6236744.
- ^ James Watson and Francis Crick (1953). «A structure for deoxyribose nucleic acid» (PDF). Nature. 171 (4356): 737–738. Bibcode:1953Natur.171..737W. doi:10.1038/171737a0. PMID 13054692. S2CID 4253007.
- ^ Crick F, Watson JD (1954). «The Complementary Structure of Deoxyribonucleic Acid». Proceedings of the Royal Society of London. 223, Series A (1152): 80–96. Bibcode:1954RSPSA.223…80C. doi:10.1098/rspa.1954.0101.
- ^ «Due credit». Nature. 496 (7445): 270. 18 April 2013. doi:10.1038/496270a. PMID 23607133.
- ^ Witkowski J (2019). «The forgotten scientists who paved the way to the double helix». Nature. 568 (7752): 308–309. Bibcode:2019Natur.568..308W. doi:10.1038/d41586-019-01176-9.
- ^ Wilkins MH, Stokes AR, Wilson HR (1953). «Molecular Structure of Deoxypentose Nucleic Acids» (PDF). Nature. 171 (4356): 738–740. Bibcode:1953Natur.171..738W. doi:10.1038/171738a0. PMID 13054693. S2CID 4280080.
- ^ Elson D, Chargaff E (1952). «On the deoxyribonucleic acid content of sea urchin gametes». Experientia. 8 (4): 143–145. doi:10.1007/BF02170221. PMID 14945441. S2CID 36803326.
- ^ Chargaff E, Lipshitz R, Green C (1952). «Composition of the deoxypentose nucleic acids of four genera of sea-urchin». J Biol Chem. 195 (1): 155–160. doi:10.1016/S0021-9258(19)50884-5. PMID 14938364.
- ^ Chargaff E, Lipshitz R, Green C, Hodes ME (1951). «The composition of the deoxyribonucleic acid of salmon sperm». J Biol Chem. 192 (1): 223–230. doi:10.1016/S0021-9258(18)55924-X. PMID 14917668.
- ^ Chargaff E (1951). «Some recent studies on the composition and structure of nucleic acids». J Cell Physiol Suppl. 38 (Suppl).
- ^ Magasanik B, Vischer E, Doniger R, Elson D, Chargaff E (1950). «The separation and estimation of ribonucleotides in minute quantities». J Biol Chem. 186 (1): 37–50. doi:10.1016/S0021-9258(18)56284-0. PMID 14778802.
- ^ Chargaff E (1950). «Chemical specificity of nucleic acids and mechanism of their enzymatic degradation». Experientia. 6 (6): 201–209. doi:10.1007/BF02173653. PMID 15421335. S2CID 2522535.
- ^ Pauling L, Corey RB (Feb 1953). «A proposed structure for the nucleic acids». Proc Natl Acad Sci U S A. 39 (2): 84–97. Bibcode:1953PNAS…39…84P. doi:10.1073/pnas.39.2.84. PMC 1063734. PMID 16578429.
- ^ «Nobel Prize — List of All Nobel Laureates».
- ^ Breslauer KJ, Frank R, Blöcker H, Marky LA (1986). «Predicting DNA duplex stability from the base sequence». PNAS. 83 (11): 3746–3750. Bibcode:1986PNAS…83.3746B. doi:10.1073/pnas.83.11.3746. PMC 323600. PMID 3459152.
- ^ Owczarzy, Richard (2008-08-28). «DNA melting temperature — How to calculate it?». High-throughput DNA biophysics. owczarzy.net. Archived from the original on 2015-04-30. Retrieved 2008-10-02.
- ^ Raq, Bio (2016). Chromosome 16: PV92 PCR Informatics Kit (1st ed.). United States: Biotechnology Explorer. p. 104.
- ^ «Chapter 9: DNA Replication – Chemistry». Retrieved 2022-06-10.
- ^ Alberts, Bruce; Johnson, Alexander; Lewis, Julian; Raff, Martin; Roberts, Keith; Walter, Peter (2002). «DNA Replication Mechanisms». Molecular Biology of the Cell. 4th Edition.
- ^ Dickerson RE (1989). «Definitions and nomenclature of nucleic acid structure components». Nucleic Acids Res. 17 (5): 1797–1803. doi:10.1093/nar/17.5.1797. PMC 317523. PMID 2928107.
- ^ Lu XJ, Olson WK (1999). «Resolving the discrepancies among nucleic acid conformational analyses». J Mol Biol. 285 (4): 1563–1575. doi:10.1006/jmbi.1998.2390. PMID 9917397.
- ^ Olson WK, Bansal M, Burley SK, Dickerson RE, Gerstein M, Harvey SC, Heinemann U, Lu XJ, Neidle S, Shakked Z, Sklenar H, Suzuki M, Tung CS, Westhof E, Wolberger C, Berman HM (2001). «A standard reference frame for the description of nucleic acid base-pair geometry». J Mol Biol. 313 (1): 229–237. doi:10.1006/jmbi.2001.4987. PMID 11601858.
- ^ Richmond; Davey, CA; et al. (2003). «The structure of DNA in the nucleosome core». Nature. 423 (6936): 145–150. Bibcode:2003Natur.423..145R. doi:10.1038/nature01595. PMID 12736678. S2CID 205209705.
- ^ Vargason JM, Eichman BF, Ho PS (2000). «The extended and eccentric E-DNA structure induced by cytosine methylation or bromination». Nature Structural Biology. 7 (9): 758–761. doi:10.1038/78985. PMID 10966645. S2CID 4420623.
- ^ Hayashi G, Hagihara M, Nakatani K (2005). «Application of L-DNA as a molecular tag». Nucleic Acids Symp Ser (Oxf). 49 (1): 261–262. doi:10.1093/nass/49.1.261. PMID 17150733.
- ^ a b Allemand JF, Bensimon D, Lavery R, Croquette V (1998). «Stretched and overwound DNA forms a Pauling-like structure with exposed bases». PNAS. 95 (24): 14152–14157. Bibcode:1998PNAS…9514152A. doi:10.1073/pnas.95.24.14152. PMC 24342. PMID 9826669.
- ^ List of 55 fiber structures Archived 2007-05-26 at the Wayback Machine
- ^ Bansal M (2003). «DNA structure: Revisiting the Watson-Crick double helix». Current Science. 85 (11): 1556–1563.
- ^ Ghosh A, Bansal M (2003). «A glossary of DNA structures from A to Z». Acta Crystallogr D. 59 (4): 620–626. doi:10.1107/S0907444903003251. PMID 12657780.
- ^ Rich A, Norheim A, Wang AH (1984). «The chemistry and biology of left-handed Z-DNA». Annual Review of Biochemistry. 53: 791–846. doi:10.1146/annurev.bi.53.070184.004043. PMID 6383204.
- ^ Sinden, Richard R (1994-01-15). DNA structure and function (1st ed.). Academic Press. p. 398. ISBN 0-12-645750-6.
- ^ Ho PS (1994-09-27). «The non-B-DNA structure of d(CA/TG)n does not differ from that of Z-DNA». Proc Natl Acad Sci USA. 91 (20): 9549–9553. Bibcode:1994PNAS…91.9549H. doi:10.1073/pnas.91.20.9549. PMC 44850. PMID 7937803.
- ^ «Double Helix». Genome.gov. Retrieved 2022-06-10.
- ^ Wing R, Drew H, Takano T, Broka C, Tanaka S, Itakura K, Dickerson R (1980). «Crystal structure analysis of a complete turn of B-DNA». Nature. 287 (5784): 755–8. Bibcode:1980Natur.287..755W. doi:10.1038/287755a0. PMID 7432492. S2CID 4315465.
- ^ Neidle, Stephen; Sanderson, Mark (2022), «DNA structure as observed in fibres and crystals», Principles of Nucleic Acid Structure, Elsevier, pp. 53–108, doi:10.1016/B978-0-12-819677-9.00007-X, ISBN 9780128196779, S2CID 239504252, retrieved 2022-06-10
- ^ Stokes, T. D. (1982). «The double helix and the warped zipper—an exemplary tale». Social Studies of Science. 12 (2): 207–240. doi:10.1177/030631282012002002. PMID 11620855. S2CID 29369576.
- ^ Gautham, N. (25 May 2004). «Response to ‘Variety in DNA secondary structure’» (PDF). Current Science. 86 (10): 1352–1353. Retrieved 25 May 2012.
However, the discovery of topoisomerases took «the sting» out of the topological objection to the plectonaemic double helix. The more recent solution of the single crystal X-ray structure of the nucleosome core particle showed nearly 150 base pairs of the DNA (i.e., about 15 complete turns), with a structure that is in all essential respects the same as the Watson–Crick model. This dealt a death blow to the idea that other forms of DNA, particularly double helical DNA, exist as anything other than local or transient structures.
[permanent dead link] - ^ Drozdetski AV, Mukhopadhyay A, Onufriev AV (November 2019). «Strongly Bent Double-Stranded DNA: Reconciling Theory and Experiment». Frontiers in Physics. 7: 195. arXiv:1907.01585. Bibcode:2019FrP…..7..195O. doi:10.3389/fphy.2019.00195. PMC 7323118. PMID 32601596.
- ^ Protozanova E, Yakovchuk P, Frank-Kamenetskii MD (2004). «Stacked–Unstacked Equilibrium at the Nick Site of DNA». J Mol Biol. 342 (3): 775–785. doi:10.1016/j.jmb.2004.07.075. PMID 15342236.
- ^ Shimada J, Yamakawa H (1984). «Ring-Closure Probabilities for Twisted Wormlike Chains. Application to DNA». Macromolecules. 17 (4): 4660–4672. Bibcode:1984MaMol..17..689S. doi:10.1021/ma00134a028.
- ^ a b Harrison RM, Romano F, Ouldridge TE, Louis AA, Doye JP (2019). «Identifying Physical Causes of Apparent Enhanced Cyclization of Short DNA Molecules with a Coarse-Grained Model». Journal of Chemical Theory and Computation. 15 (8): 4660–4672. doi:10.1021/acs.jctc.9b00112. PMC 6694408. PMID 31282669.
- ^ Travers, Andrew (2005). «DNA Dynamics: Bubble ‘n’ Flip for DNA Cyclisation?». Current Biology. 15 (10): R377–R379. doi:10.1016/j.cub.2005.05.007. PMID 15916938. S2CID 10568179.
- ^ Konrad MW, Bolonick JW (1996). «Molecular dynamics simulation of DNA stretching is consistent with the tension observed for extension and strand separation and predicts a novel ladder structure». Journal of the American Chemical Society. 118 (45): 10989–10994. doi:10.1021/ja961751x.
- ^ Roe DR, Chaka AM (2009). «Structural basis of pathway-dependent force profiles in stretched DNA». Journal of Physical Chemistry B. 113 (46): 15364–15371. doi:10.1021/jp906749j. PMID 19845321.
- ^ Bosaeus N, Reymer A, Beke-Somfai T, Brown T, Takahashi M, Wittung-Stafshede P, Rocha S, Nordén B (2017). «A stretched conformation of DNA with a biological role?». Quarterly Reviews of Biophysics. 50: e11. doi:10.1017/S0033583517000099. PMID 29233223.
- ^ Taghavi A, van Der Schoot P, Berryman JT (2017). «DNA partitions into triplets under tension in the presence of organic cations, with sequence evolutionary age predicting the stability of the triplet phase». Quarterly Reviews of Biophysics. 50: e15. doi:10.1017/S0033583517000130. PMID 29233227.
- ^ Crick FH (1976). «Linking numbers and nucleosomes». Proc Natl Acad Sci USA. 73 (8): 2639–43. Bibcode:1976PNAS…73.2639C. doi:10.1073/pnas.73.8.2639. PMC 430703. PMID 1066673.
- ^ Prunell A (1998). «A topological approach to nucleosome structure and dynamics: the linking number paradox and other issues». Biophys J. 74 (5): 2531–2544. Bibcode:1998BpJ….74.2531P. doi:10.1016/S0006-3495(98)77961-5. PMC 1299595. PMID 9591679.
- ^ Luger K, Mader AW, Richmond RK, Sargent DF, Richmond TJ (1997). «Crystal structure of the nucleosome core particle at 2.8 A resolution». Nature. 389 (6648): 251–260. Bibcode:1997Natur.389..251L. doi:10.1038/38444. PMID 9305837. S2CID 4328827.
- ^ Davey CA, Sargent DF, Luger K, Maeder AW, Richmond TJ (2002). «Solvent mediated interactions in the structure of the nucleosome core particle at 1.9 Å resolution». Journal of Molecular Biology. 319 (5): 1097–1113. doi:10.1016/S0022-2836(02)00386-8. PMID 12079350.
Статья на конкурс «био/мол/текст»: ДНК — двойная спираль? Не всегда. Отдельные островки наших молекул наследственности могут по ошибке принимать довольно экзотические формы. Например, сворачиваться в спирали из четырех полигуаниновых нитей — вопреки классическим принципам молекулярной биологии. Но действительно ли подобные аномалии возникают «по ошибке»? Или природа давно уже «оседлала» эту странность нуклеиновых кислот, поставив её себе на службу? Можно ли считать четверные G-спирали рабочими «деталями» сложнейшей машины геномной регуляции? И случайна ли их причастность к процессам старения и канцерогенеза?
В мистическом фильме Д. Аронофски «Фонтан» присутствует весьма интересный образ. Конкистадор, отправившийся по велению испанской королевы на поиски древа вечной жизни (далекими прототипами героев здесь, по-видимому, послужили первооткрыватель Флориды Понсе де Леон и король Испании Фердинанд), находит его в храме индейцев Майя в Южной Америке. Однако, вкусив млечного сока этого древа, герой понимает, что что-то пошло не так. Вместо того чтобы обрести бессмертие, он начинает прорастать цветущей травой и полностью становится субстратом для этой буйной, паразитической по сути, растительности. Иными словами, у человека и у природы могут быть разные, если можно так выразиться, представления о жизненной силе и долголетии. И, возможно, именно поэтому в поисках путей продления жизни, мы постоянно натыкаемся на опасность канцерогенеза. Будь это исследования в области стволовых клеток, попытки преодолеть так называемый предел Хейфлика с помощью фермента теломеразы либо иные способы борьбы с клеточным старением. Всякий раз геронтология и онкология идут «рука об руку», теснейшим образом переплетаясь. И одной из точек сопряжения этих двух областей можно считать удивительные структурные аномалии ДНК, носящие название G-квадруплексов…
ДНК не по канону
Мы привыкли думать о ДНК как о двойной спирали, в которой азотистые основания нуклеотидов на противоположных цепях однозначно соответствуют друг другу: аденин — тимину, гуанин — цитозину. Эта, безусловно, фундаментальная особенность нуклеиновых кислот лежит в основе механизмов наследственности. Именно благодаря ей становятся возможными удвоение и корректирование ДНК, а также реализация генетической информации в структуре РНК и белков.
Однако на деле наши молекулы наследственности оказываются куда гибче, подвижнее и причудливее, нежели это было некогда описано легендарными нобелевскими лауреатами Дж. Уотсоном и Ф. Криком. Например, подобно РНК, ДНК может формировать так называемые шпильки, которые на двойной спирали приобретают вид крестообразных структур (рис. 1). Эти «аномальные» образования принимают активное участие в регуляции работы с генетической информацией и задействованы как в копировании ДНК (репликации) [1], так и в переносе информации с ДНК на РНК (транскрипции) [2].

Рисунок 1. Формирование крестоообразных структур ДНК. Рисунок с сайта www.physoc.org.
Конечно, «крестами» и шпильками дело не ограничивается. К числу возможных конформаций ДНК относятся также тройные (рис. 2Б) и даже четверные спирали (рис. 2В), возникающие в результате неканонических связей между азотистыми основаниями [3]. В течение последнего десятилетия заметно возросло внимание к так называемым G-квадруплексам — структурам, представляющим собой спирали из четырех нитей ДНК или РНК, соединенных одними только гуанинами. Постепенно выясняется, что эти образования играют весьма важную роль в регуляции активности генов, в генетической изменчивости и в функционировании теломер [4].

Рисунок 2. Двойная (а), тройная (б) и четверная (в) спирали ДНК. Рисунок с сайта www.konan-fiber.jp.

Рисунок 3. «G-ДНК». а. Строение G-тетрады; М+ — одновалентный катион. б. Формирование G-квадруплекса из 1) одной, 2) двух и 3) четырех нитей ДНК. Рисунок с сайта www.chem.cmu.edu.
Способность гуанина к самоассоциации была обнаружена еще в конце 19 века. И только в 1962 году удалось установить, что в растворах он образует агрегаты из четырех молекул (называемых G-квартетом, или G-тетрадой) [5]. Такие тетрады скрепляются между собой неканоническими (то есть, не предусмотренными в модели Уотсона — Крика) водородными связями, называемыми «хугстиновскими» — по фамилии их первооткрывателя [6]. При этом входящие в них гуанины располагаются в одной плоскости и нуждаются в стабилизации моновалентными катионами (например, K+ или Na+) (рис. 3а).
Содержащие гуанин нуклеиновые кислоты в растворе могут образовывать такие структуры из одной, двух или четырех различных нитей (рис. 3б). Однако стабильными они будут лишь в том случае, когда три и более G-тетрады сгруппируются в плотную «стопку», «подперев» друг друга межплоскостными стекинг-взаимодействиями и сформировав тем самым G-квадруплекс (G4-структуру). А для этого, в свою очередь, необходима «счастливая встреча» четырех полигуаниновых участков (GGGn), находящихся либо на одной, либо на разных молекулах ДНК или РНК [7].
Правда, на каждое правило есть свои исключения. Здесь можно привести в пример синдром ломкой X-хромосомы, возникающий вследствие экспансии многочисленных (более 200) повторов (CGG) в гене FMR1, необходимом для развития нервной системы. Матричная РНК такого мутантного гена способна формировать вполне стабильные четверные спирали даже из двухгуаниновых мотивов. То есть, в нашей «стопке» будет всего лишь две G-тетрады (рис. 4) [10].
«Значит, это кому-нибудь нужно?» © В. Маяковский

Рисунок 4. «Двуслойный» квадруплекс, формирующийся из четырех мотивов (GG) на РНК гена FMR1 при синдроме ломкой X-хромосомы. Рисунок с сайта www.computer.org.
Разумеется, то, что возможно в пробирке, далеко не всегда имеет место в живой клетке. Однако последние годы было убедительно показано, что G-квадруплексы действительно образуются in vivo в хромосомах живых организмов [11, 12]. И, кроме того, методами биоинформатики в геноме человека обнаружено порядка 376 тысяч районов, в которых потенциально могут возникать эти четырехспиральные образования. Интересно, что такие участки, как правило, находятся в составе регуляторных генетических элементов (промоторов, терминаторов, нетранслируемых последовательностей матричной РНК), теломер, рибосомных РНК и интронов, то есть последовательностей, не кодирующих структуру белка. Тогда как кодирующие фрагменты ДНК (экзоны) преимущественно «очищены» от подобных нуклеотидных мотивов [13, 14]. И неудивительно! Ведь, забегая вперед, нельзя не отметить, что такие гуаниновые «узелки» способны становиться очагами генетической нестабильности, провоцируя появление серьезных мутаций. Поэтому направленность естественного отбора в данном случае вполне понятна.
Однако почему-то всё тот же естественный отбор способствовал заметному скоплению потенциальных G4-структур в области генных промоторов, то есть, участков ДНК, определяющих производительность данного гена и позволяющих её регулировать [13]. Причем особенно ярко это выражено в группе онкогенов, повышенная активность которых необходима для возникновения и развития раковых опухолей. Тогда как, к примеру, встречаемость вероятных G-квадруплексов в промоторах генов — супрессоров раковых опухолей (призванных подавлять канцерогенез) значительно ниже, чем в среднем по геному [15, 16]. Более того, последовательности многих промоторов и интронов, особо обогащенных потенциальными G4-структурами, достаточно консервативны, то есть несут общие черты у целого ряда эукариотических организмов [17, 18].
Картину дополняет то обстоятельство, что живые системы не пожалели времени и сил на выработку специальных белков, способных связываться с G-квадруплексами, либо формируя и стабилизируя их, либо «разворачивая», «расплетая» или просто разрезая [19]. Бесполезным или «не существующим» в клетке структурам (каковыми их вплоть до недавнего времени считали многие исследователи) эволюция столько внимания не уделяет. Как отметил более 30 лет назад нобелевский лауреат Аарон Клуг: «Если G-квадруплексы так легко формируются in vitro, природа, должно быть, нашла путь применения их in vivo» [4]. С каждым годом мы получаем всё больше подтверждений этому прозорливому высказыванию.
Из двойной спирали — в четверную
Чтобы сформировать в ДНК такую крупную структуру, как G-квадруплекс, необходимо предварительно подвергнуть плавлению (разъединить) соответствующий участок классической двойной спирали. Длина таких участков может составлять несколько десятков нуклеотидов. К примеру, общая формула для поиска потенциальных G4-структур в геноме, использованная в работах исследователей из Кэмбриджского университета, выглядела как (G3+N1—7G3+N1—7G3+N1—7G3) — то есть, предполагала длину квадруплекса от 15 до 33 пар оснований [13] (хотя это далеко не единственный вариант [14]). Теоретически G-квадруплексы могут возникнуть практически в любом месте генома— при условии образования достаточно длинного однонитевого ДНК-фрагмента [16]. Другое дело, что по показателям стабильности они будут существенно проигрывать.
Наиболее благоприятные условия для высвобождения протяженных кусочков одноцепочечной ДНК создаются, прежде всего, во время репликации. И действительно: количество G4-структур в клетке заметно (приблизительно в пять раз) возрастает именно при удвоении хромосом [20]. В идеале, по завершении копирования наследственного материала все гуаниновые «узелки» (и старые, и новые) должны удаляться, поскольку в это время клетка буквально «причесывает» свою ДНК. Однако, как показывает практика, часть G-квадруплексов остается и присутствует в геноме на протяжении всего клеточного цикла [11, 12, 20]. Более того, по-видимому, сам процесс репликации нуждается в наличии G4-структур. В частности, они входят в состав ориджинов (геномных районов, с которых начинается репликация) позвоночных животных и, по одной из версий, определяют направление движения ДНК-полимеразы — «копировальной машины» ДНК [21, 22].
Образованию четверных гуаниновых спиралей могут способствовать не только репликация, но и транскрипция (считывание гена), а также репарация (починка) хромосом [23]. Кроме того, в ядре возможно спонтанное плавление двойной спирали ДНК, возникающее в результате тех или иных молекулярных эффектов [24, 25]. В конце концов, появление G4-структур может быть обусловлено целенаправленным воздействием на ДНК специальных белков — шаперонов, призванных формировать квадруплексы там, где это положено [19].
Куда легче дело обстоит с РНК, однонитевыми по своей природе молекулами. Исключительная гибкость и отсутствие «обременения» в виде второй комплементарной цепочки позволяет им свободно принимать самые разнообразные конформации. И не случайно потенциальные G4-спирали нашлись в нетранслируемых (то есть, входящих в состав матричной РНК, но структуру белка не кодирующих) областях более чем 3000 человеческих генов [26, 27]. Кроме того, в последние годы перспективным представляется исследование гибридных квадруплексов, состоящих из ДНК и РНК. Интересно, что для их формирования достаточно не четырех, а всего лишь двух полигуаниновых участков на ДНК (два других автоматически будут присутствовать на считываемой с гена РНК) [28].
На самом краю хромосомы
А теперь — всё внимание таким удивительным ядерным структурам, как теломеры [29]! Располагаясь на концах линейных хромосом ядерных организмов, они призваны защищать генетический материал от разрушения. Структуры всех эукариотических теломер удивительно консервативны: они представляют собой цепочку из многократно повторяющихся нуклеотидных мотивов. В человеческом геноме это мотив (TTAGGG) (обратим внимание читателя на тройной гуанин), воспроизведенный 1000–2000 раз [30]! Не правда ли, идеальная среда для возникновения многочисленных G-квадруплексов? Но и это еще не всё: на конце теломер человека и других теплокровных животных располагается довольно протяженный, 30–300 нуклеотидов, участок однонитевой ДНК со свободным 3’-концом [31]. Состоит он из всё тех же повторов (ТТАGGG). И уж здесь-то образованию гуаниновых узелков ничто не мешает.
Теломерные последовательности, будучи извлеченными из ядра или синтезированными, действительно массово формируют G-квадруплексы [4]. Однако доказательство их наличия в клетке требует куда более тонких подходов. С помощью меченых антител, специфически связывающихся с G4-структурами, было показано, что последние действительно образуются на концах хромосом (правда, не всех). По крайней мере, около 20% всех сигналов были зафиксированы именно там [20]. Возникают ли обнаруженные квадруплексы собственно в теломерах либо в близлежащих областях хромосом, по этим данным сказать трудно, поскольку разрешение метода сильно ограничено. Однако нам достоверно известно, что многие теломерные белки действительно взаимодействуют с G4-структурами (либо формируя их, либо разрушая), и это один из главных аргументов в пользу их полноценного участия в жизни теломер [19]. А уж о том, что эта «жизнь» чрезвычайно важна для нас в контексте вопроса о здоровье и долголетии, говорить не приходится.
Судя по всему, G-квадруплексы задействованы в формировании защитного колпачка, или кэпа, на конце теломеры. В клетках человека такое «кэпирование» осуществляется шелтерином — комплексом из шести белков. Он предохраняет линейную хромосому от излишнего внимания клеточных «контролёров», считающих, что конец цепочки ДНК — это повреждение, требующее соответствующих методов «лечения». А в области теломер такое «лечение» оборачивается катастрофическими последствиями: «сшивкой» разных хромосом и, в итоге, гибелью клетки либо запуском канцерогенеза [32].
С G4-структурами взаимодействуют такие шелтериновые белки, как TRF2 и POT1. При этом TRF2 связывает и стабилизирует квадруплексы на однонитевом 3’-фрагменте, тогда как POT1 обусловливает распад гуаниновых четверных спиралей [19]. Иными словами, квадруплексы при образовании «кэпа» нужны, но дозированно.
Предполагаются самые разнообразные варианты участия G4 ДНК в «кэпировании» хромосом [33]. G-квадруплекс может быть необходим для образования специфической теломерной структуры, именуемой T-петлей (теломерной петлей) (рис. 5, 6а). Он возникает либо при простом запетливании однонитевого 3’-фрагмента (рис. 6б), либо при его внедрении в двойную спираль теломерной ДНК (рис. 6в) [34]. Таким образом осуществляется маскировка свободного 3’-конца (в общем случае распознаваемого клеткой как сигнал тревоги) [33].

Рисунок 5. Т-петля на конце теломеры под электронным микроскопом. Фото с сайта web.pdx.edu.

Рисунок 6. Различные виды Т-петель. а. T-петля без квадруплекса; б. T-петля с квадруплексом, замыкающим однонитевой 3’-фрагмент теломеры на себя; в. T-петля, образующая квадруплекс при внедрении однонитевого 3’-конца в двойную спираль теломерной ДНК. Рисунок с сайта pubs.rsc.org.
Помимо этого, обсуждаются альтернативные T-петле способы «кэпирования» хромосом. По одной из версий, одноцепочечная 3’-ДНК формирует плотный ряд G-квадруплексов, стабилизирующих однонитевой фрагмент и предохраняющих его от деградации особо ретивыми клеточными белками (рис. 7) [35]. Вероятно, подобный способ защиты теломеры используется в тех случаях, когда полноценного шелтерина на конце хромосомы не образуется [36].

Рисунок 7. Защита теломеры с помощью серии G-квадруплексов, образующихся на однонитевом 3’-конце. Рисунок из [4].
Не исключено, что в клетках человека в той или иной мере реализуются оба способа «обороны» теломеры с участием G4-образований. Иное дело, что такая «оборона» может оборачиваться для нас рядом неприятных моментов.
Точка обратного отсчета
Согласно наиболее популярной точке зрения, именно в теломерах спрятан ключ (по крайней мере, один из главных таких ключей) к пониманию процесса старения и ограниченной продолжительности нашей жизни. Линейные хромосомы, в отличие от циклических, коими могут похвастаться большинство доядерных организмов, как правило, не способны удваиваться вечно. Каждый цикл репликации, в силу особенностей организации этого ферментативного механизма, приводит к укорочению одной из дочерних нитей ДНК на 50–100 нуклеотидов. В итоге, после 50–60 раундов копирования наследственного материала потери в длине теломеры становятся неприемлемыми, поэтому клетка запускает процесс своего старения (сенесценции) либо гибнет [30, 37]. Правда, всем известные стволовые клетки являются ярким исключением из этого общего правила [38, 39]. (А еще — клетки растений, меняющие длину теломер в зависимости от времени года [40].)
Исследуя механизмы старения человека, невозможно обойти вниманием такое наследственное заболевание как синдром Вернера. Проявляется оно в ускоренном старении организма и сопровождается всеми сопутствующими эффектами: снижением иммунитета, повышением риска раковых заболеваний, многочисленными физиологическими нарушениями, характерными для людей преклонного возраста. Причиной этого синдрома оказалось нарушение структуры белка — геликазы WRN, основной функцией которого является расплетание теломерных квадруплексов. В результате во время репликации синтез дочерней цепи ДНК тормозится и обрывается в месте образования G4-структуры. Это приводит к галопирующему укорочению теломер и, соответственно, раннему запуску клеточного старения [41]. Аналогичного эффекта в культурах клеток можно добиться искусственным путем, повышая стойкость квадруплексов за счет их связывания со специальными химическими веществами [42].
Можно спорить по поводу применимости модели наследственного заболевания в деле расшифровки механизмов нормального, не патологичного старения. Однако совершенно не исключено, что не расплетенные вовремя теломерные квадруплексы задействованы и в этом случае. Вспомним, к примеру, как долгое время считалось, что синдром прогерии Хатчинсона-Гилфорда, связанный с преждевременным старением у детей, не имеет отношения к старению как таковому. В дальнейшем же было показано, что вызывающий эту болезнь прогерин образуется и в коже обычных пожилых людей [43].
Помимо этого, не секрет, что одной из вероятных причин старения является накопление в геноме повреждений, вызванных окислением свободными радикалами [44]. Причем существенная часть из них концентрируется именно на теломерах [45]. Возможных объяснений такому предпочтительному поражению кончиков хромосом довольно много. По-видимому, в этом виновны и сниженная (силами шелтериновых белков) активность ферментов «ремонта» ДНК, и особенности нуклеотидной последовательности теломерной ДНК. И здесь опять имеет смысл вспомнить про наши G-квадруплексы! Ведь они, помимо всего прочего, оказываются особенно чувствительными к окислительным повреждениям [46].
Смертельно опасная «вечная» жизнь
Говоря о теломерных G4-структурах, нельзя обойти вниманием тот интригующий факт, что, располагаясь на концах 3’-выступа, они блокируют работу теломеразы. А ведь это тот самый широко популяризованный фермент, на который возлагалось столько надежд, и за изучение которого в 2009 году была вручена Нобелевская премия по физиологии и медицине! Теломераза наращивает 3’-концы теломер и таким образом нивелирует их естественное укорочение. Иными словами — продлевает активную жизнь хромосомам и, соответственно, клетке [29]. Однако G-квадруплексы не просто мешают теломеразе делать свое дело — помимо этого, они, располагаясь в области промотора гена TERT, подавляют ее синтез в клетке [47]. И, конечно, этот эффект можно было бы счесть за негативный, если бы не один весьма печальный факт. Дело в том, что избыточная активность теломеразы создает весьма благоприятную среду для перерождения нормальных клеток в раковые. От работы этого фермента сильно зависят порядка 90% всех злокачественных опухолей, ведь он позволяет перерожденным клеткам делиться бесконечно долго [47].
Блокирование работы теломеразы составляет одну из функций гена BRCA1. BRCA1 — широко известный супрессор раковых опухолей, то есть ген — «борец» с канцерогенезом. Он является важным компонентом системы исправления ошибок в ДНК и контролирует активность целого ряда генов, в том числе задействованных в образовании опухолей [48]. Поэтому мутации в BRCA1 зачастую приводят к развитию рака груди и яичников [49], а в России они — в «лидерах» генетических причин возникновения онкозаболеваний [50]. Интересно, что подавление теломеразы этим (весьма немаловажным, как мы убедились) белком осуществляется не только за счет регуляции активности кодирующего её гена TERT [51], но и за счет прямого вмешательства в её работу на теломерах. В частности, предполагают, что BRCA1 связывается с теломерными квадруплексами и стабилизирует их, в результате чего наш «фермент молодости» остаётся не у дел [19].
Как было отмечено выше, TERT — отнюдь не единственный регулируемый G-квадруплексами ген, благоприятствующий развитию злокачественных опухолей [15]. Помимо него в этом ряду фигурируют c-MYC, c-KIT, BLC2, VEGF, HIF-1a и целый ряд других известных онкогенов [52]. Разумеется, такая закономерность не могла не привлечь внимания ученых, занятых поиском средств против рака. Сегодня существует целый ряд работ, посвященных влиянию тех или иных квадруплекс-связывающих веществ на рост и развитие раковых клеток. И, по результатам этих исследований, искусственное укрепление G-квадруплексов представляется чрезвычайно перспективным путем лечения онкологических заболеваний [47, 52].
По лезвию бритвы
Как гласит известная легенда, Будда Шакьямуни обрел просветление, услышав фразу учителя музыки, адресованную его ученику: «Если ты ослабишь струну, музыка не зазвучит, а если ты перетянешь её, она порвется». И этот принцип в полной мере относится ко множеству, если не ко всем биологическим процессам.
Те же самые G4-структуры, которые столь перспективны в рамках лечения раковых опухолей, могут сами по себе становиться провокаторами канцерогенных заболеваний. Вспомним вышеупомянутый синдром преждевременного старения Вернера. Возникает он на фоне недееспособности геликазы WRN, в норме «развязывающей» гуаниновые «узелки» на теломерах. В наших клетках существует целый спектр таких белков, призванных тем или иным способом удалять G-квадруплексы, дабы освободить дорогу ферментам репликации. Если же этого не происходит, то копирование ДНК стопорится в месте образования G4-структуры. В результате запускается целый ряд процессов, приводящих к разрывам ДНК, хромосомным перестройкам и мутациям наследственного материала [36, 41].
Так, например, при повреждении гена специализированной на G-квадруплексах геликазы BLM возникает синдром Блума. Проявляется он в низкорослости и ранней предрасположенности к целому ряду онкозаболеваний. Нарушение функции геликазы FANJ приводит к развитию анемии Фалькони, сопровождающейся повышенной хрупкостью хромосом и склонностью к лейкозам. Аналогичным образом мутации в гене геликазы RETL1 увеличивают чувствительность организма к некоторым видам рака. Этот список можно было бы продолжить целым рядом наследственных заболеваний, провоцирующих канцерогенез. И все они связаны с генетической и теломерной нестабильностью, вызванной «стойкими» ДНК- и РНК-квадруплексами [41].
Иными словами, чтобы «музыка», исполняемая оркестром генных и теломерных G-квадруплексов, звучала, необходима тонкая настройка их жесткости. По-видимому, мы имеем дело с чрезвычайно широкой и сложной сетью структурных ДНК-аномалий. Возникающих как спонтанно, так и при помощи специальных белков. Чувствительных как к молекулярному окружению, так и к физико-химическим условиям среды. Способных как предотвращать раковые заболевания, так и провоцировать их. Принимающих участие как в защите теломер, так и в процессах клеточного старения.
- Pearson C.E., Zorbas H., Price G.B., Zannis-Hadjopoulos M. (1996). Inverted repeats, stem-loops, and cruciforms: significance for initiation of DNA replication. J. Cell Biochem. 63, 1–22;
- Wadkins R.M. (2000). Targeting DNA secondary structures. Curr. Med. Chem. 7, 1–15;
- Франк-Каменецкий М. (2015). Необычные формы ДНК. Портал «ПостНаука»;
- Rhodes D. and Lipps H.J. (2015). G-quadruplexes and their regulatory roles in biology. Nucl. Acids Res. 43, 8627–8637;
- Gellert M., Lipsett M.N., Davies D.R. (1962). Helix formation by guanylic acid. Proc. Natl. Acad. Sci. USA. 48, 2013–2018;
- Hoogsteen K. (1963). The crystal and molecular structure of a hydrogen-bonded complex between 1-methylthymine and 9-methyladenine. Acta Cryst. 16, 907–916;
- Burge S., Parkinson G.N., Hazel P., Todd A.K, Neidle S. (2006). Quadruplex DNA: sequence, topology and structure. Nucl. Acids Res. 34, 5402–5415;
- Kotlyar A.B., Borovok N., Molotsky T., Cohen H., Shapir E., Porath D. (2005). Long, monomolecular guanine-based nanowires. Adv. Mat. 17, 1901–1905;
- Ngo V.A., Felice R.D., Haas S. (2014). Is the G-quadruplex an effective nanoconductor for ions? J. Phys. Chem. B. 118, 864–872;
- Kettania A., Kumara A.R., Patela D.J. (2014). Solution structure of a DNA quadruplex containing the fragile X syndrome triplet repeat. J. Mol. Biol. 254, 638–656;
- Lam E.Y., Beraldi D., Tannahill D., Balasubramanian S. (2013). G-quadruplex structures are stable and detectable in human genomic DNA. Nat. Commun. 4, 1796–1780;
- Rodriguez R., Miller K.M., Forment J.V., Bradshaw C.R., Nikan M., Britton S. et al. (2012). Small-molecule-induced DNA damage identifies alternative DNA structures in human genes. Nat. Chem. Biol. 8, 301–310;
- Huppert J.L. and Balasubramanian S. (2005). Prevalence of quadruplexes in the human genome. Nucl. Acids Res. 33, 2908–2916;
- Todd A.K., Johnston M., Neidle S. (2005). Highly prevalent putative quadruplex sequence motifs in human DNA. Nucl. Acids Res. 33, 2901–2907;
- Eddy J. and Maizels N. (2006). Gene function correlates with potential for G4 DNA formation in the human genome. Nucl. Acids Res. 34, 3887–3896;
- Maizels N. and Gray L.T. (2013). The G4 Genome. PLoS Genet. 9, e1003468;
- Fernando H., Reszka A.P., Huppert J., Ladame S., Rankin S., Venkitaraman A.R. et al. (2006). A conserved quadruplex motif located in a transcription activation site of the human c-kit oncogene. Biochem. 45, 7854–7860;
- Eddy J. and Maizels N. (2008). Conserved elements with potential to form polymorphic G-quadruplex structures in the first intron of human genes. Nucl. Acids Res. 36, 1321–1333;
- Brazda V., Haronikova L., Liao J.C., Fojta M. (2014). DNA and RNA quadruplex-binding proteins. Int. J. Mol. Sci. 15, 17493–17517;
- Biffi G., Tannahill D., McCafferty J., Balasubramanian S. (2013). Quantitative visualization of DNA G-quadruplex structures in human cells. Nat. Chem. 5, 182–186;
- Leonard A.C. and Mechali M. (2013). DNA replication origins. Cold Spring Harb. Perspect. Biol. 5, a010116;
- Valton A.L., Hassan-Zadeh V., Lema I., Boggetto N., Alberti P., Saintome C. et al. (2014). G4 motifs affect origin positioning and efficiency in two vertebrate replicators. EMBO J. 33, 732–746;
- Li W., Wu P., Ohmichi T., Sugimoto N. (2002). Characterization and thermodynamic properties of quadruplex/duplex competition. FEBS Lett. 526, 77–81;
- Miyoshi D., Karimata H., Sugimoto N. (2006). Hydration regulates thermodynamics of G-quadruplex formation under molecular crowding conditions. J. Am. Chem. Soc. 128, 7957–7963;
- Nikolova E.N., Kim E., Wise A.A., O’Brien P.J., Andricioaei I., Al-Hashimi H.M. (2011). Transient Hoogsteen base pairs in canonical duplex DNA. Nature. 470, 498–502;
- Bugaut A. and Balasubramanian S. (2012). 5′-UTR RNA G-quadruplexes: translation regulation and targeting. Nucl. Acids Res. 40, 4727–4741;
- Beaudoin J.D. and Perreault J.P. (2013). Exploring mRNA 3′-UTR G-quadruplexes: evidence of roles in both alternative polyadenylation and mRNA shortening. Nucl. Acids Res. 41, 5898–5911;
- Xiao S., Zhang J.Y., Zheng K.W., Hao Y.H., Tan Z. (2013). Bioinformatic analysis reveals an evolutional selection for DNA:RNA hybrid G-quadruplex structures as putative transcription regulatory elements in warm-blooded animals. Nucl. Acids Res. 41, 10379–10390;
- «Нестареющая» Нобелевская премия: в 2009 году отмечены работы по теломерам и теломеразе;
- Старение — плата за подавление раковых опухолей?;
- Makarov V.L., Hirose Y., Langmore J.P. (1997). Long G tails at both ends of human chromosomes suggest a C strand degradation mechanism for telomere shortening. Cell. 88, 657–666;
- Sfeir A. and Lange T. (2012). Removal of shelterin reveals the telomere end-protection problem. Science. 336, 593–597;
- Galati A., Micheli E., Cacchione S. (2013). Chromatin Structure in Telomere Dynamics. Front. Oncol. 3, 46;
- Табу в науке: лаборатория, задававшая неправильные вопросы;
- Bochman M.L., Paeschke K., Zakian V.A. (2012). DNA secondary structures: stability and function of G-quadruplex structures. Nat. Rev. Genet. 13, 770–780;
- Brosh R.M. Jr. (2011). Put on your thinking cap: G-quadruplexes, helicases, and telomeres. Aging (Albany NY). 3, 332–335;
- Levy M.Z., Allsopp R.Z., Futcher A.B., Greider C.W., Harley C.B. (1992). Telomere end-replication problem and cell aging. J. Mol. Biol. 225, 951–960;
- Была клетка простая, стала стволовая;
- Ствол и ветки: стволовые клетки;
- Длина теломер и времена года;
- Brosh R.M. Jr. (2013). DNA helicases involved in DNA repair and their roles in cancer. Nat. Rev. Cancer. 13, 542–558;
- Incles C.M., Schultes C.M., Kempski H., Koehler H., Kelland L.R., Neidle S. (2004). A G-quadruplex telomere targeting agent produces p16-associated senescence and chromosomal fusions in human prostate cancer cells. Mol. Cancer Ther. 3, 1201;
- Burtner C.R. and Kennedy B.K. (2010). Progeria syndromes and ageing: what is the connection? Nat. Rev. Mol. Cell Biol. 11, 567–578;
- Wickens A.P. (2001). Ageing and the free radical theory. Respir. Physiol. 128, 379–391;
- Sun L., Tan R., Xu J., LaFace J., Gao Y., Xiao Y. et al. (2015). Targeted DNA damage at individual telomeres disrupts their integrity and triggers cell death. Nucl. Acids Res. 43, 6334–6347;
- Clark D.W., Phang T., Edwards M.G., Geraci M.W., Gillespie M.N. (2012). Promoter G-quadruplex sequences are targets for base oxidation and strand cleavage during hypoxia-induced transcription. Free Radic. Biol. Med. 53, 51–59;
- Patel D.J., Phan A.T., Kuryavyi V. (2007). Human telomere, oncogenic promoter and 5′-UTR G-quadruplexes: diverse higher order DNA and RNA targets for cancer therapeutics. Nucl. Acids Res. 35, 7429–7455;
- Rosen E.M. (2013). BRCA1 in the DNA damage response and at telomeres. Front. Genet. 4, 85;
- Рак молочной железы с семейной историей;
- Любченко Л.Н. Наследственный рак молочной железы и/или яичников: ДНК-диагностика, индивидуальный прогноз, лечение и профилактика: дис. … д-ра мед. наук. — Москва, 2009;
- Xiong J., Fan S., Meng Q., Schramm L., Wang C., Bouzahza B. et al. (2003). BRCA1 inhibition of telomerase activity in cultured cells. Mol. Cell Biol. 23, 8668–8690;
- Balasubramanian S., Hurley L.H., Neidle S. (2011). Targeting G-quadruplexes in gene promoters: a novel anticancer strategy? Nat. Rev. Drug Discov. 10, 261–275..